38重InDel快速族群推断体系的优化及验证
王庆国 , 1 , 2 , * , 赵蕾 , 2 , * , 李唐松 3 , 付旺 4 , 谢何鑫 2 , 马原 3 , 孙文平 , 1 , 韩俊萍 , 2 , 3
1.山东第一医科大学第一附属医院(山东省千佛山医院)病理科,山东 济南 250014
2.公安部物证鉴定中心 北京市现场物证检验工程技术研究中心 现场物证溯源技术国家工程实验室,北京 100038
3.北京市公安局朝阳分局刑事侦查支队,北京 100038
4.北京市上地医院,北京 100193
Optimization and Developmental Validation of 38-plex InDel Panel for Ancestry Inference
WANG Qing-guo , 1 , 2 , * , ZHAO Lei , 2 , * , LI Tang-song 3 , FU Wang 4 , XIE He-xin 2 , MA Yuan 3 , SUN Wen-ping , 1 , HAN Jun-ping , 2 , 3
1.First Affiliated Hospital of Shandong First Medical University (Shandong Provincial Qianfoshan Hospital), Jinan 250014, China
2.National Engineering Laboratory for Forensic Science, Beijing Engineering Research Center of Crime Scene Evidence Examination, Institute of Forensic Science, Beijing 100038, China
3.Detachment of Criminal Investigation, Chaoyang Branch of Beijing Public Security Bureau, Beijing 100038, China
4.Beijing Shangdi Hospital, Beijing 100193, China
通讯作者: 孙文平,男,副教授,主要从事肿瘤病理学和法医学研究;E-mail:wenpingsun@163.com 韩俊萍,女,博士,副主任法医师,主要从事法医遗传学研究;E-mail:pgww19861025@163.com
第一联系人: 王庆国和赵蕾为共同第一作者
编委: 刘希玲
收稿日期: 2021-01-15
基金资助:
国家科技资源共享服务平台计划资助项目 . YCZYPT[2017]01-3 基本科研业务费专项资金资助项目 . 2017JB025
摘要
目的 对构建的38重InDel快速族群推断体系进行优化,根据DNA分析方法科学工作组(Scientific Working Group on DNA Analysis Method,SWGDAM)指南进行性能验证,并对其应用于东亚、欧洲、非洲及其混合人群族群推断的准确性进行验证。 方法 以DNA标准品9947A为模板,通过调整引物平衡性、Mg2+ 终浓度、优化PCR热循环参数和扩增体积等建立最优扩增条件,比较样本的等位基因丢失、非特异性扩增以及推断样本来源是否与已知信息匹配,评价该体系的相关性能。 结果 本体系的最佳模板用量为0.125~2 ng DNA,InDel分型结果准确,扩增均衡性好,种属特异性好;对混有血红蛋白(≤80 µmol/L)、靛蓝(≤40 mmol/L)、钙离子(≤1.0 mmol/L)以及腐殖酸(≤90 ng/µL)等抑制剂的样本具有一定耐受性;可以直接扩增检测血卡、唾液卡,且族群推断结果准确;能够区分两样本的混合DNA样本;对实际案例常见生物检材的检验结果良好。 结论 38重InDel快速族群推断体系分型结果准确可靠,体系性能符合SWGDAM指南要求,可以准确推断未知个体的非洲、欧洲、东亚及欧亚混合人群来源,可用于法医鉴定实践。
关键词:
法医遗传学 族群推断 插入/缺失多态性 复合扩增 性能验证
Abstract
Objective The previously established 38-plex InDel system was optimized and its performance was validated according to the Scientific Working Group on DNA Analysis Method (SWGDAM) application guidelines. The ancestry inference accuracy of individuals from East Asian, European, African and mixed populations was verified. Methods DNA standard sample 9947A was used as the template to establish the optimal amplification conditions by adjusting primer balance, Mg2+ final concentration and optimizing PCR thermal cycle parameters and amplification volume. The allelic dropout, nonspecific amplification and whether the origin of the inferred samples matched the known information were compared to evaluate the performance of this system. Results The optimal dosage of this system was 0.125-2 ng DNA template. The results of InDel typing were accurate, the amplification equilibrium was good, and the species specificity was good. This system showed certain tolerance to DNA samples including the inhibitor such as hemoglobin (≤80 μmol/L), indigo (≤40 mmol/L), calcium ion (≤1.0 mmol/L), and humic acid (≤90 ng/μL). The system enabled the direct amplification of DNA from saliva and blood on filter paper, and the results of ethnic inference were accurate. The system successfully detected the mixed DNA sample from two individuals. The test results of the system for common biological materials in practical cases were accurate. Conclusion The results of the 38-plex InDel system are accurate and reliable, and the performance of the system meets the requirement of the SWGDAM guidelines. This system can accurately differentiate the ancestry origins of individuals from African, European, East Asian, and Eurasian populations and can be implemented in forensic practice.
Keywords:
forensic genetics ancestry inference InDel polymorphism multiplex amplification developmental validation
本文引用格式
王庆国, 赵蕾, 李唐松, 付旺, 谢何鑫, 马原, 孙文平, 韩俊萍. 38重InDel快速族群推断体系的优化及验证 . 法医学杂志 [J], 2022, 38(5): 611-617 DOI:10.12116/j.issn.1004-5619.2021.510105
WANG Qing-guo, ZHAO Lei, LI Tang-song, FU Wang, XIE He-xin, MA Yuan, SUN Wen-ping, HAN Jun-ping. Optimization and Developmental Validation of 38-plex InDel Panel for Ancestry Inference . Journal of Forensic Medicine 10.12116/j.issn.1004-5619.2021.510105
目前个体识别DNA鉴定,主要是对现场生物检材进行短串联重复序列聚合酶链反应(short tandem repeat-polymerase chain reaction,STR-PCR)分析,利用STR分型数据与数据库或嫌疑人进行比对,以达到同一认定的目的,当比对失败时,案件则会陷入被动。祖先信息标记(ancestry informative mark,AIM)是指在不同地域人群之间具有等位基因频率差异的标记,筛选一组相应的AIM构建复合扩增体系,通过检测未知样本的基因分型,推断未知个体的祖先成分与遗传结构,实现不同洲际或不同地域人群的有效区分[1 -5 ] 。
常用的AIM有单核苷酸多态性(single nucleotide polymorphisms,SNP)和插入/缺失(insertion-deletion,InDel)突变遗传标记。InDel遗传标记广泛存在于人类基因组(约占5%),包含一个或多个核苷酸插入或缺失所构成的片段长度多态性[6 ] 。InDel兼具SNP和STR的优点,检测片段短,适合降解DNA的检测。此外,因具有片段长度差异,故可采用目前法医DNA实验室通用的毛细管电泳(capillary electrophoresis,CE)平台分型检测,易于推广,InDel复合扩增体系可以通过等位基因峰高比值判别混合样本[7 -10 ] 。
本研究针对前期[11 ] 构建的38重InDel快速族群推断体系(包含38个InDel遗传标记、1个性别基因座Amelogenin 和1个Y-STR基因座)进行优化,根据DNA分析方法科学工作组(Scientific Working Group on DNA Analysis Method,SWGDAM)指南验证其准确性、灵敏度、重复性、种属特异性、耐受性、对混合样本和实际案例检材的检测能力以及直接扩增能力等性能,以评估该体系的法医学应用价值。
1 材料与方法
1.1 数据和样本
38重InDel快速族群推断体系的参考数据库包含来自25个人群的2 450份个体的分型数据[11 ] ;马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔的DNA共10份,由公安部物证鉴定中心提取并定量;28例案例样本(血斑5例、脱落细胞9例、精斑5例、烟蒂6例、唾液斑3例);95份无关个体的血卡(70份)和唾液卡(25份)及2份新疆回族样本(XJH287和XJH291);已知分型的DNA样本(9947A、9948和志愿者LCX)均由公安部物证鉴定中心提供。所有样本对象均签署知情同意书。本研究已通过公安部物证鉴定中心伦理委员会的伦理审查(审批号2017—004)。
1.2 DNA提取和定量
采用QIAamp DNA Blood Midi试剂盒(德国Qiagen公司)提取上述样本DNA,使用NanoDrop 2000c分光光度计(美国Thermo Scientific公司)进行定量。以去离子灭菌水调整质量浓度至1~2 ng/μL备用。
1.3 38重InDel族群推断体系的优化
1.3.1 引物平衡调整
38个遗传标记引物基本信息(染色体位置、特定SNP的识别号码、荧光标记、扩增子大小)见文献[11 ],先等比混合制成引物混合物(起始浓度均为50 µmol/L),采用试剂盒推荐的体系及扩增程序进行实验,即10 µL反应体系包括2.5×NiHi S9 PCR Mix[PCR预混液,内含镁离子(Mg2+ )5 mmol/L,苏州新海生物科技股份有限公司]4 µL,引物混合物1 µL,模板DNA 1 µL(质量浓度1~2 ng/µL),灭菌水4 µL。扩增程序:95 ℃ 10 min;95 ℃ 10 s,58 ℃ 1 min,共28个循环;60 ℃ 30 min,25 ℃保温。取1 µL PCR产物与9 µL甲酰胺、内标AS500(L)(苏州新海生物科技股份有限公司)混匀,95 ℃ 3 min后立即冰浴5 min。CE在3130xl 基因分析仪(美国Applied Biosystems公司)上进行,使用性能最优的高分子聚合物7号(performance optimized polymer 7,POP7)凝胶,36 cm毛细管进行电泳分离。使用GeneMapper® ID v3.2软件(美国Thermo Fisher Scientific公司)进行分型。根据各个位点等位基因荧光信号强度的高低,调整各基因座引物配比浓度,直至38个位点全部扩出且峰高较为均衡,最终确定各位点引物浓度。
1.3.2 Mg2+ 终浓度的筛选
分别向反应体系中添加Mg2+ 0、0.2、0.4、0.6 µL(Mg2+ 初始浓度为25 mmol/L),即10 µL反应体系的终浓度分别为2、2.5、3、3.5 mmol/L,参照1.3.1节建立10 µL反应体系和扩增程序。同时设置阴性对照。实验重复3次。
1.3.3 PCR扩增条件优化
循环数:将循环次数设为26、28、30、32次,其他步骤同1.3.1节。
退火温度:根据引物的DNA熔解温度(melting temperature,Tm)设置退火温度梯度,将退火温度设为54 ℃、56 ℃、58 ℃、60 ℃、62 ℃,按照上述最佳循环数设置,其他步骤同1.3.1节。
终延伸时间:将终延伸反应时间设为10、20、40、60 min,按照上述最佳循环数和退火/延伸温度设置,其他步骤同1.3.1节。
1.3.4 反应体积
将反应体积设为5、10、25、50 µL,体系配制见表1 。采用1.3.3节优化的PCR条件,对比4种反应体积的扩增效果,选择效果最佳者作为PCR的反应体积。
1.4 38重InDel族群推断体系的性能验证
准确性:取DNA标准品9947A、9948(苏州新海生物科技股份有限公司)以及随机取2份志愿者DNA样本(XJH287和XJH291)对其38个InDel位点进行Sanger法测序[由生工生物工程(上海)股份有限公司测序],以验证本体系检测结果的准确性。
灵敏度:以去离子灭菌水将DNA标准品9947A稀释至5、2、1、0.5、0.25、0.125、0.062 5、0.031 25 ng,按本体系方法检测,平行重复3次。
重复性:从95份无关个体的血卡、唾液卡中随机取20份样本,由公安部物证鉴定中心和北京市公安局朝阳分局实验室同时按本体系方法进行扩增和分型检测,设置9947A为阳性对照,去离子水代替DNA为阴性对照。两家实验室分别采用3130xl 和3500xL基因分析仪(美国Applied Biosystems公司)进行电泳分离。
种属特异性:分别取马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔的DNA,按本体系方法进行扩增并检测,平行重复3次。
耐受性:在1 ng 9947A的PCR反应体系中,第一组分别加入血红蛋白(美国Sigma公司),使其终浓度为60、80、100、150、200 µmol/L;第二组分别加入靛蓝(美国Sigma公司),使其终浓度为30、40、50、60、70 mmol/L;第三组分别加入钙离子(美国Sigma公司),使其终浓度为0.8、1、1.25、1.5、2 µmol/L;第四组分别加入腐殖酸(美国Sigma公司),使其终浓度为80、90、100、130、160 ng/µL,按本体系方法检测。
混合样本分析:选取2种已知分型的DNA样本(标准品9947A和志愿者LCX),分别稀释至1 ng/µL备用,作为单一样本分型结果的阳性对照,将LCX样本作为混合样本分析中的主要贡献者成分设置混合比例梯度,在保证DNA总模板用量为1 ng的基础上,使反应体系中9947A与LCX的体积呈1∶1、1∶2、1∶4、1∶9的比例,按本体系方法检测,平行重复3次。
案例样本适用性:28例模拟案例样本采用本体系方法进行扩增与检测。
直接扩增能力:利用本体系对70份无关个体的血卡和25份唾液卡进行直接扩增,血卡或唾液卡用直径为0.5 mm的手持式打孔器打下一圆片,加入反应体系中,扩增体系和条件与1.3节中筛选的相同。
1.5 数据分析
采用本体系方法所得InDel分型图谱,获得34个以上位点(除Amelogenin 和DYS439 )分型正确且各等位基因峰高大于50相对荧光单位(relative fluorescent unit,RFU)为有效分型。使用项目组自主研发的族群推断软件DNA Ancestry Analyzer v1.0(简称DAA,公安部物证鉴定中心研制)计算样本的人群祖先成分比例、群体的Pm和似然比(likelihood ratio,LR)。参照文献[11 ]方法对样本来源进行判断,并与样本已知来源信息比较。
2 结果
2.1 Mg2+ 终浓度对扩增效果的影响
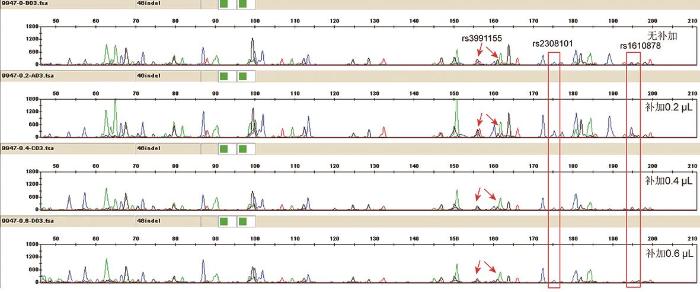
结果(图1 )显示:当反应体系中Mg2+ 的终浓度为3.0、3.5 mmol/L时,出现rs3991155位点丢失;当反应体系Mg2+ 的终浓度为2.5 mmol/L时,扩增产物的峰高值略高于终浓度为2 mmol/L时,且杂合子均衡性更好,如rs2308101位点,故后续实验反应体系中Mg2+ 终浓度为2.5 mmol/L,即补加0.2 µL Mg2+ 。
图1
图1
Mg2+ 不同终浓度条件下9947A产物的CE图谱
箭头表示位点丢失;红色框表示均衡性变化。
Fig. 1
CE maps from amplification of 9947A with different concentrations of Mg2+
2.2 PCR条件优化
2.2.1 循环数
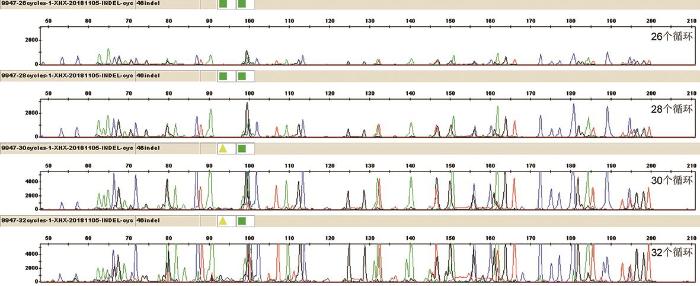
采用10 µL体系优化循环数,结果显示,循环数为26~32次时均可得到完整的InDel分型结果,产物信号强度随着循环数增加而增强(图2 ),由于26次循环时产物峰值很低,而30、32次循环时产物信号过强且出现非特异峰,因此选择28次循环作为PCR循环数。
图2
图2
不同循环条件下9947A产物的CE图谱
Fig. 2
CE maps from amplification of 9947A with different cycle numbers
2.2.2 退火温度
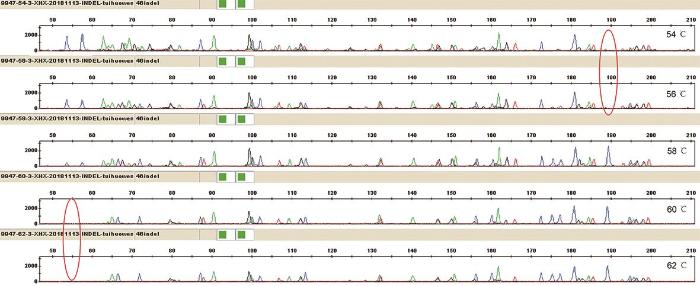
结果显示,54 ℃和56 ℃时rs16416位点出现等位基因丢失,60 ℃和62 ℃时rs3054057位点出现等位基因丢失,58 ℃时可以得到完整InDel分型结果(图3 ),因此选择58 ℃作为退火温度。
图3
图3
不同退火温度条件下9947A产物的CE图谱
红色圆圈表示位点丢失。
Fig. 3
CE maps from amplification of 9947A with different annealing temperatures
2.2.3 终延伸时间
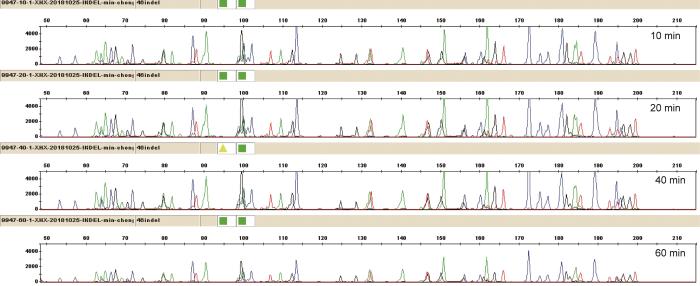
采用10 µL体系优化终延伸时间,结果显示,终延伸时间为10~60 min时均未出现等位基因丢失且扩增效率相似(图4 ),考虑缩短整个反应时间,故选择10 min作为终延伸时间。
图4
图4
不同终延伸时间条件下9947A产物的CE图谱
Fig. 4
CE maps from amplification of 9947A with different final extension times
经上述PCR条件优化,完成整个PCR扩增检测系统显示时间仅需63 min。
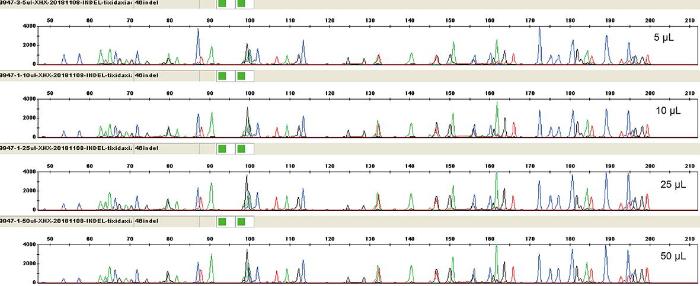
2.2.4 反应体积
通过比较4个反应体积的扩增结果,发现4种体积下均能获得完整InDel分型(图5 ),但5 µL体积下的整体信号强度低于其他3种体积,为了不影响分型效果同时降低实验成本,故选择10 µL作为反应终体积。
图5
图5
不同反应体系条件下9947A产物的CE图谱
Fig. 5
CE maps from amplification of 9947A with different reaction volumes
2.3 准确性
比对9947A、9948、XJH287和XJH291 4份DNA样本的Sanger测序结果与本体系检测结果,其分型完全一致,表明本体系对38个InDel位点的基因分型结果准确性可靠。
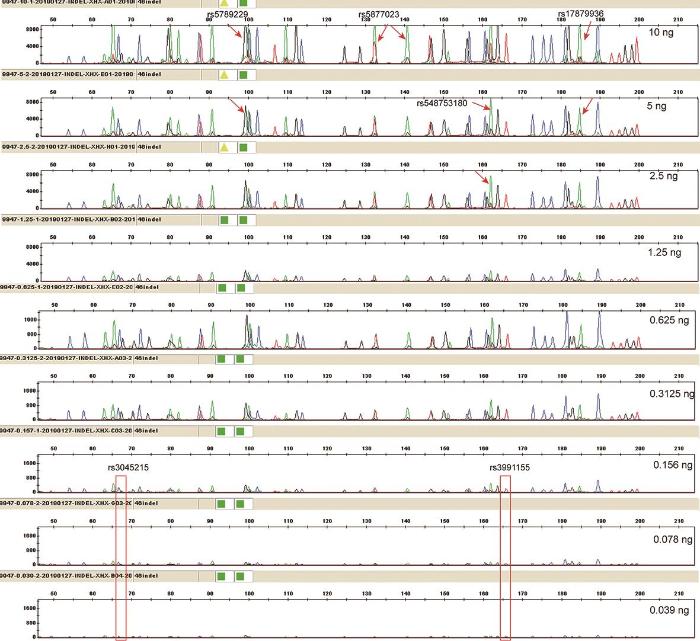
2.4 灵敏度
利用模板量在0.031 25~5 ng的DNA标准品9947A,进行体系的灵敏度测试,结果表明,当DNA模板量在5 ng时,rs1130852位点产物荧光信号过强,荧光信号扩散到其他荧光通道,干扰分型结果的判定(图6 );当DNA模板量为0.062 5 ng时,可获得38个位点的完整分型,但产物峰值偏低;当DNA模板量为0.031 25 ng时,出现rs3991155和rs67625332位点丢失(图6 )。故本体系的最低检测限为0.062 5 ng,最佳DNA模板用量为0.125~2 ng。
图6
图6
9947A的灵敏度检测结果
箭头表示荧光渗透;红色框表示位点丢失。
Fig. 6
The sensitivity study results of 9947A
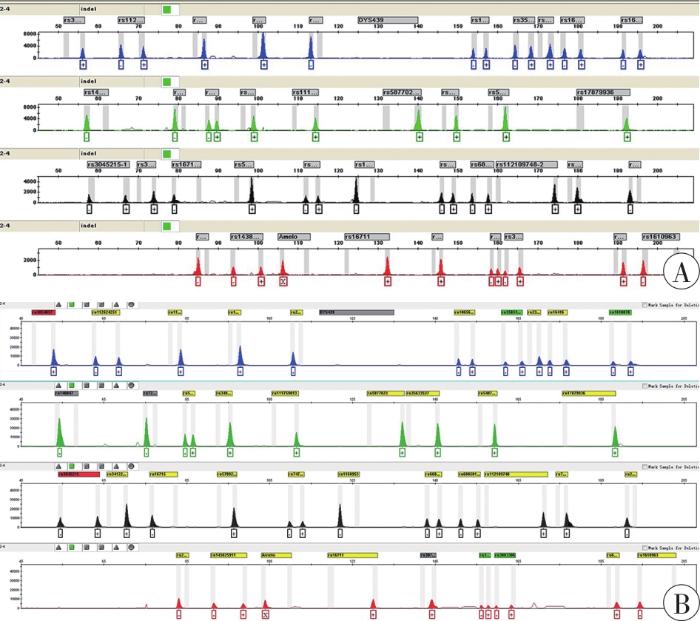
2.5 重复性
20份DNA样本按照本体系方法,由两家单位分别进行检测,因遗传分析仪型号及毛细管使用次数的差异,个别位点产物信号强度有所差异(图7 ),经比对,20份样本的InDel分型结果完全一致,说明本体系具有较好的重复性。
图7
图7
2家实验室使用38重InDel快速族群推断体系检测1个样本的结果比对
A:某样本在公安部物证鉴定中心实验室的检验结果;B:某样本在朝阳分局实验室的检验结果。
Fig. 7
Comparison of test results of a sample from two laboratories using the 38-plex InDel assay
2.6 种属特异性
通过马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔共10种非人源的DNA进行检测,同时检测9947A标准品,结果显示,马和牛检测出个别片段,但均不在设置的等位基因bin图谱上,其与人来源9947A的检测结果有明显区别,表明本体系种属特异性良好,可以有效区分人源样本与非人源样本。
2.7 耐受性
在含DNA标准品9947A的扩增体系中,分别加入4种不同浓度的血红蛋白、靛蓝、钙离子和腐殖酸,检验结果(表2 )显示,当血红蛋白≤80 µmol/L、靛蓝≤40 mmol/L、钙离子≤1.0 mmol/L、腐殖酸≤90 ng/µL时,可得到完整的InDel分型结果,而高于上述浓度则出现不同程度的等位基因丢失,甚至得不到分型结果。
2.8 混合样本分析
随着9947A∶LCX的体积按1∶1、1∶2、1∶4、1∶9混合比例增加,rs3054057位点的峰高不均衡性逐渐增大,同时rs5877023、rs16416也观察到类似现象。当混合比例为1∶4时,9947A作为次要贡献者成分,虽然信号值较低,但仍可获得完整的分型结果;当混合比例为1∶9时,优势扩增过于明显,次要贡献者成分9947A出现大量等位基因的丢失。
2.9 案例样本适用性
28例模拟案例样本经本体系方法检验,除1份脱落细胞样本因出现大量等位基因丢失而未得到有效分型(常规STR检验也未获得分型)外,其余样本均可获得完整的InDel分型结果。
2.10 直接扩增能力
结果显示,1份唾液卡出现大量等位基因丢失导致未获得有效分型,其余94份样本均获得有效分型,经DAA软件分析,所有样本的族群推断结果均与样本已知信息一致,准确率达100%。
3 讨论
InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用。InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断。目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] 。2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分。2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分。
本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性。除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] 。同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示。后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况。
根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致。与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短。与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测。
综上,本研究对已建立的38重InDel快速族群推断体系进行优化,同时验证了该体系的各项性能指标,结果表明该体系的性能符合SWGDAM指南要求,能够在65 min内获得准确InDel分型并可实现三大洲际族群地域来源推断,可在法医实践中选择使用。
参考文献
View Option
[1]
DE LA PUENTE M SANTOS C FONDEVILA M et al The global AIMs nano set: A 31-plex SNaPshot assay of ancestry- informative SNPs
[J]. Forensic Sci Int Genet ,2016 ,22 :81 -88 . doi:10.1016/j.fsigen.2016.01.015 .
[本文引用: 1]
[2]
FONDEVILA M PHILLIPS C SANTOS C et al Revision of the SNPforID 34-plex forensic ancestry test: Assay enhancements, standard reference sample genotypes and extended population studies
[J]. Forensic Sci Int Genet ,2013 ,7 (1 ):63 -74 . doi:10.1016/j.fsigen.2012.06.007 .
[3]
PHILLIPS C SALAS A SÁNCHEZ J J et al Inferring ancestral origin using a single multiplex assay of ancestry-informative marker SNPs
[J]. Forensic Sci Int Genet ,2007 ,1 (3/4 ):273 -280 . doi:10.1016/j.fsigen.2007.06.008 .
[4]
LI C X PAKSTIS A J JIANG L et al A panel of 74 AISNPs: Improved ancestry inference within Eastern Asia
[J]. Forensic Sci Int Genet ,2016 ,23 :101 -110 . doi:10.1016/j.fsigen.2016.04.002 .
[5]
WEI Y L WEI L ZHAO L et al A single-tube 27-plex SNP assay for estimating individual ancestry and admixture from three continents
[J]. Int J Leg Med ,2016 ,130 (1 ):27 -37 . doi:10.1007/s00414-015-1183-5 .
[本文引用: 2]
[6]
PEREIRA R PHILLIPS C ALVES C et al A new multiplex for human identification using insertion/deletion polymorphisms
[J]. Electrophoresis ,2009 ,30 (21 ):3682 -3690 . doi:10.1002/elps.200900274 .
[本文引用: 2]
[7]
PEREIRA R PHILLIPS C PINTO N et al Straightforward inference of ancestry and admixture proportions through ancestry-informative insertion deletion multiplexing
[J]. PLoS One ,2012 ,7 (1 ): e29684 . doi:10.1371/journal.pone.0029684 .
[本文引用: 4]
[8]
FONDEVILA M PHILLIPS C SANTOS C et al Forensic performance of two insertion-deletion marker assays
[J]. Int J Leg Med ,2012 ,126 (5 ):725 -737 . doi:10.1007/s00414-012-0721-7 .
[本文引用: 1]
[9]
SANTOS C PHILLIPS C OLDONI F et al Completion of a worldwide reference panel of samples for an ancestry informative Indel assay
[J]. Forensic Sci Int Genet ,2015 ,17 :75 -80 . doi:10.1016/j.fsigen.2015.03.011 .
[10]
ZAUMSEGEL D ROTHSCHILD M A SCHNEIDER P M A 21 marker insertion deletion polymorphism panel to study biogeographic ancestry
[J]. Forensic Sci Int Genet ,2013 ,7 (2 ):305 -312 . doi:10.1016/j.fsigen.2012.12.007 .
[本文引用: 3]
[11]
[本文引用: 5]
HAN J P ZHAO L WANG Q G et al A single-tube direct amplification 38-plex ancestry informative InDels assay and its integration with a microfluidic system
[J]. Shengwu Huaxue Yu Shengwu Wuli Jinzhan ,2020 ,47 (9 ):968 -981 .
[本文引用: 5]
[12]
MILLS R E LUTTIG C T LARKINS C E et al An initial map of insertion and deletion (INDEL) variation in the human genome
[J]. Genome Res ,2006 ,16 (9 ):1182 -1190 . doi:10.1101/gr.4565806 .
[本文引用: 1]
[13]
BASTOS-RODRIGUES L PIMENTA J R PENA S D J The genetic structure of human populations studied through short insertion-deletion polymorphisms
[J]. Ann Hum Genet ,2006 ,70 (Pt 5 ):658 -665 . doi:10.1111/j.1469-1809.2006.00287.x .
[本文引用: 1]
[14]
LAN Q SHEN C M JIN X Y et al Front Cover: Distinguishing three distinct biogeographic regions with an in-house developed 39-AIM-InDel panel and further admixture proportion estimation for Uyghurs
[J]. Electrophoresis ,2019 ,40 (11 ):elps. 201970101 . doi:10.1002/elps.201970101 .
[本文引用: 2]
[15]
[本文引用: 1]
JIANG L SUN Q F MA Q et al Optimization and validation of analysis method based on 27-plex SNP panel for ancestry inference
[J]. Yichuan ,2017 ,39 (2 ):166 -173 .
[本文引用: 1]
[16]
PARK S J KIM J Y YANG Y G et al Direct STR amplification from whole blood and blood- or saliva-spotted FTA without DNA purification
[J]. J Forensic Sci ,2008 ,53 (2 ):335 -341 . doi:10.1111/j.1556-4029.2008.00666.x .
[本文引用: 1]
[17]
SANTOS C FONDEVILA M BALLARD D et al Forensic ancestry analysis with two capillary electrophoresis ancestry informative marker (AIM) panels: Results of a collaborative EDNAP exercise
[J]. Forensic Sci Int Genet ,2015 ,19 :56 -67 . doi:10.1016/j.fsigen.2015.06.004 .
[本文引用: 1]
[18]
解通 35个InDel位点荧光复合扩增检测体系的构建、验证及新疆三个少数民族遗传背景的探索研究
[D].广州 :南方医科大学 ,2019 :76 -86 .
[本文引用: 2]
XIE T Development and validation of 35 InDels fluorescence multiplex system and genetic background explorative analysis of three ethnic minorities in Chinese Xinjiang area
[D]. Guangzhou : Southern Medical University ,2019 :76 -86 .
[本文引用: 2]
[19]
ZHUANG B HAN J P XIANG G X et al A fully integrated and automated microsystem for rapid pharmacogenetic typing of multiple warfarin-related single-nucleotide polymorphisms
[J]. Lab Chip ,2016 ,16 (1 ):86 -95 . doi:10.1039/c5lc01094b .
[本文引用: 1]
The global AIMs nano set: A 31-plex SNaPshot assay of ancestry- informative SNPs
1
2016
... 目前个体识别DNA鉴定,主要是对现场生物检材进行短串联重复序列聚合酶链反应(short tandem repeat-polymerase chain reaction,STR-PCR)分析,利用STR分型数据与数据库或嫌疑人进行比对,以达到同一认定的目的,当比对失败时,案件则会陷入被动.祖先信息标记(ancestry informative mark,AIM)是指在不同地域人群之间具有等位基因频率差异的标记,筛选一组相应的AIM构建复合扩增体系,通过检测未知样本的基因分型,推断未知个体的祖先成分与遗传结构,实现不同洲际或不同地域人群的有效区分[1 -5 ] . ...
Revision of the SNPforID 34-plex forensic ancestry test: Assay enhancements, standard reference sample genotypes and extended population studies
0
2013
Inferring ancestral origin using a single multiplex assay of ancestry-informative marker SNPs
0
2007
A panel of 74 AISNPs: Improved ancestry inference within Eastern Asia
0
2016
A single-tube 27-plex SNP assay for estimating individual ancestry and admixture from three continents
2
2016
... 目前个体识别DNA鉴定,主要是对现场生物检材进行短串联重复序列聚合酶链反应(short tandem repeat-polymerase chain reaction,STR-PCR)分析,利用STR分型数据与数据库或嫌疑人进行比对,以达到同一认定的目的,当比对失败时,案件则会陷入被动.祖先信息标记(ancestry informative mark,AIM)是指在不同地域人群之间具有等位基因频率差异的标记,筛选一组相应的AIM构建复合扩增体系,通过检测未知样本的基因分型,推断未知个体的祖先成分与遗传结构,实现不同洲际或不同地域人群的有效区分[1 -5 ] . ...
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
A new multiplex for human identification using insertion/deletion polymorphisms
2
2009
... 常用的AIM有单核苷酸多态性(single nucleotide polymorphisms,SNP)和插入/缺失(insertion-deletion,InDel)突变遗传标记.InDel遗传标记广泛存在于人类基因组(约占5%),包含一个或多个核苷酸插入或缺失所构成的片段长度多态性[6 ] .InDel兼具SNP和STR的优点,检测片段短,适合降解DNA的检测.此外,因具有片段长度差异,故可采用目前法医DNA实验室通用的毛细管电泳(capillary electrophoresis,CE)平台分型检测,易于推广,InDel复合扩增体系可以通过等位基因峰高比值判别混合样本[7 -10 ] . ...
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
Straightforward inference of ancestry and admixture proportions through ancestry-informative insertion deletion multiplexing
4
2012
... 常用的AIM有单核苷酸多态性(single nucleotide polymorphisms,SNP)和插入/缺失(insertion-deletion,InDel)突变遗传标记.InDel遗传标记广泛存在于人类基因组(约占5%),包含一个或多个核苷酸插入或缺失所构成的片段长度多态性[6 ] .InDel兼具SNP和STR的优点,检测片段短,适合降解DNA的检测.此外,因具有片段长度差异,故可采用目前法医DNA实验室通用的毛细管电泳(capillary electrophoresis,CE)平台分型检测,易于推广,InDel复合扩增体系可以通过等位基因峰高比值判别混合样本[7 -10 ] . ...
... InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用.InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断.目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] .2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
... [7 ]从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
Forensic performance of two insertion-deletion marker assays
1
2012
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
Completion of a worldwide reference panel of samples for an ancestry informative Indel assay
0
2015
A 21 marker insertion deletion polymorphism panel to study biogeographic ancestry
3
2013
... 常用的AIM有单核苷酸多态性(single nucleotide polymorphisms,SNP)和插入/缺失(insertion-deletion,InDel)突变遗传标记.InDel遗传标记广泛存在于人类基因组(约占5%),包含一个或多个核苷酸插入或缺失所构成的片段长度多态性[6 ] .InDel兼具SNP和STR的优点,检测片段短,适合降解DNA的检测.此外,因具有片段长度差异,故可采用目前法医DNA实验室通用的毛细管电泳(capillary electrophoresis,CE)平台分型检测,易于推广,InDel复合扩增体系可以通过等位基因峰高比值判别混合样本[7 -10 ] . ...
... InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用.InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断.目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] .2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
单管直扩38重祖先信息InDels体系的构建及其在微流控芯片系统的应用
5
2020
... 本研究针对前期[11 ] 构建的38重InDel快速族群推断体系(包含38个InDel遗传标记、1个性别基因座Amelogenin 和1个Y-STR基因座)进行优化,根据DNA分析方法科学工作组(Scientific Working Group on DNA Analysis Method,SWGDAM)指南验证其准确性、灵敏度、重复性、种属特异性、耐受性、对混合样本和实际案例检材的检测能力以及直接扩增能力等性能,以评估该体系的法医学应用价值. ...
... 38重InDel快速族群推断体系的参考数据库包含来自25个人群的2 450份个体的分型数据[11 ] ;马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔的DNA共10份,由公安部物证鉴定中心提取并定量;28例案例样本(血斑5例、脱落细胞9例、精斑5例、烟蒂6例、唾液斑3例);95份无关个体的血卡(70份)和唾液卡(25份)及2份新疆回族样本(XJH287和XJH291);已知分型的DNA样本(9947A、9948和志愿者LCX)均由公安部物证鉴定中心提供.所有样本对象均签署知情同意书.本研究已通过公安部物证鉴定中心伦理委员会的伦理审查(审批号2017—004). ...
... 38个遗传标记引物基本信息(染色体位置、特定SNP的识别号码、荧光标记、扩增子大小)见文献[11 ],先等比混合制成引物混合物(起始浓度均为50 µmol/L),采用试剂盒推荐的体系及扩增程序进行实验,即10 µL反应体系包括2.5×NiHi S9 PCR Mix[PCR预混液,内含镁离子(Mg2+ )5 mmol/L,苏州新海生物科技股份有限公司]4 µL,引物混合物1 µL,模板DNA 1 µL(质量浓度1~2 ng/µL),灭菌水4 µL.扩增程序:95 ℃ 10 min;95 ℃ 10 s,58 ℃ 1 min,共28个循环;60 ℃ 30 min,25 ℃保温.取1 µL PCR产物与9 µL甲酰胺、内标AS500(L)(苏州新海生物科技股份有限公司)混匀,95 ℃ 3 min后立即冰浴5 min.CE在3130xl 基因分析仪(美国Applied Biosystems公司)上进行,使用性能最优的高分子聚合物7号(performance optimized polymer 7,POP7)凝胶,36 cm毛细管进行电泳分离.使用GeneMapper® ID v3.2软件(美国Thermo Fisher Scientific公司)进行分型.根据各个位点等位基因荧光信号强度的高低,调整各基因座引物配比浓度,直至38个位点全部扩出且峰高较为均衡,最终确定各位点引物浓度. ...
... 采用本体系方法所得InDel分型图谱,获得34个以上位点(除Amelogenin 和DYS439 )分型正确且各等位基因峰高大于50相对荧光单位(relative fluorescent unit,RFU)为有效分型.使用项目组自主研发的族群推断软件DNA Ancestry Analyzer v1.0(简称DAA,公安部物证鉴定中心研制)计算样本的人群祖先成分比例、群体的Pm和似然比(likelihood ratio,LR).参照文献[11 ]方法对样本来源进行判断,并与样本已知来源信息比较. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
单管直扩38重祖先信息InDels体系的构建及其在微流控芯片系统的应用
5
2020
... 本研究针对前期[11 ] 构建的38重InDel快速族群推断体系(包含38个InDel遗传标记、1个性别基因座Amelogenin 和1个Y-STR基因座)进行优化,根据DNA分析方法科学工作组(Scientific Working Group on DNA Analysis Method,SWGDAM)指南验证其准确性、灵敏度、重复性、种属特异性、耐受性、对混合样本和实际案例检材的检测能力以及直接扩增能力等性能,以评估该体系的法医学应用价值. ...
... 38重InDel快速族群推断体系的参考数据库包含来自25个人群的2 450份个体的分型数据[11 ] ;马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔的DNA共10份,由公安部物证鉴定中心提取并定量;28例案例样本(血斑5例、脱落细胞9例、精斑5例、烟蒂6例、唾液斑3例);95份无关个体的血卡(70份)和唾液卡(25份)及2份新疆回族样本(XJH287和XJH291);已知分型的DNA样本(9947A、9948和志愿者LCX)均由公安部物证鉴定中心提供.所有样本对象均签署知情同意书.本研究已通过公安部物证鉴定中心伦理委员会的伦理审查(审批号2017—004). ...
... 38个遗传标记引物基本信息(染色体位置、特定SNP的识别号码、荧光标记、扩增子大小)见文献[11 ],先等比混合制成引物混合物(起始浓度均为50 µmol/L),采用试剂盒推荐的体系及扩增程序进行实验,即10 µL反应体系包括2.5×NiHi S9 PCR Mix[PCR预混液,内含镁离子(Mg2+ )5 mmol/L,苏州新海生物科技股份有限公司]4 µL,引物混合物1 µL,模板DNA 1 µL(质量浓度1~2 ng/µL),灭菌水4 µL.扩增程序:95 ℃ 10 min;95 ℃ 10 s,58 ℃ 1 min,共28个循环;60 ℃ 30 min,25 ℃保温.取1 µL PCR产物与9 µL甲酰胺、内标AS500(L)(苏州新海生物科技股份有限公司)混匀,95 ℃ 3 min后立即冰浴5 min.CE在3130xl 基因分析仪(美国Applied Biosystems公司)上进行,使用性能最优的高分子聚合物7号(performance optimized polymer 7,POP7)凝胶,36 cm毛细管进行电泳分离.使用GeneMapper® ID v3.2软件(美国Thermo Fisher Scientific公司)进行分型.根据各个位点等位基因荧光信号强度的高低,调整各基因座引物配比浓度,直至38个位点全部扩出且峰高较为均衡,最终确定各位点引物浓度. ...
... 采用本体系方法所得InDel分型图谱,获得34个以上位点(除Amelogenin 和DYS439 )分型正确且各等位基因峰高大于50相对荧光单位(relative fluorescent unit,RFU)为有效分型.使用项目组自主研发的族群推断软件DNA Ancestry Analyzer v1.0(简称DAA,公安部物证鉴定中心研制)计算样本的人群祖先成分比例、群体的Pm和似然比(likelihood ratio,LR).参照文献[11 ]方法对样本来源进行判断,并与样本已知来源信息比较. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
An initial map of insertion and deletion (INDEL) variation in the human genome
1
2006
... InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用.InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断.目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] .2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
The genetic structure of human populations studied through short insertion-deletion polymorphisms
1
2006
... InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用.InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断.目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] .2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
Front Cover: Distinguishing three distinct biogeographic regions with an in-house developed 39-AIM-InDel panel and further admixture proportion estimation for Uyghurs
2
2019
... InDel遗传标记因具有片段长度差异,故可与常规STR检验方法相同,采用聚合酶链反应-毛细管电泳(polymerase chain reaction-capillary electrophoresis,PCR-CE)技术进行分型检测[12 ] ,易于在国内法医DNA实验室推广应用.InDel遗传标记在不同人群间可以表现出等位基因频率的差异,故可针对不同人群选择相应的位点构建体系,基于检测样本的位点分型,对其祖先成分和遗传结构进行分析,实现个体的生物地理起源推断.目前,国内外已有多种基于InDel遗传标记的相关研究,可用于不同洲际或不同地域人群的区分[7 ,10 ,13 -14 ] .2012年,PEREIRA等[7 ] 从相关文献报道及Marshfield数据库中筛选出46个AIM InDel位点构建复合扩增体系,所选位点等位基因频率在非洲、欧洲、东亚及美洲人群间的差异值≥0.4,通过对HGDP-CEPH数据库参考人群样本以及Bele’m市的226名混合人群样本的分析,表明该组位点可用于非洲、欧洲、东亚及美洲人群的区分,并可评估混合人群的遗传成分.2019年,LAN等[14 ] 构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
... [14 ]构建了1套包含39个InDel位点的复合扩增体系,利用1000 Genomes中15个人群1 511名个体的分型数据,对所选位点的族群推断效能进行初步评估,证明了该体系可以实现三大洲际人群(东亚、欧洲、非洲)的有效区分. ...
27-plex SNP种族推断方法的优化及验证
1
2017
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
27-plex SNP种族推断方法的优化及验证
1
2017
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
Direct STR amplification from whole blood and blood- or saliva-spotted FTA without DNA purification
1
2008
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
Forensic ancestry analysis with two capillary electrophoresis ancestry informative marker (AIM) panels: Results of a collaborative EDNAP exercise
1
2015
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
35个InDel位点荧光复合扩增检测体系的构建、验证及新疆三个少数民族遗传背景的探索研究
2
2019
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
35个InDel位点荧光复合扩增检测体系的构建、验证及新疆三个少数民族遗传背景的探索研究
2
2019
... 本实验室前期基于38个InDel遗传标记构建了单管复合扩增体系,可有效进行东亚、欧洲、非洲三大人群以及混合人群的遗传结构分析和个体的祖先来源推断,相较于27-Plex SNP检测体系[5 ,15 ] ,本体系为直接扩增体系,可采用0.5 mm直径打孔器从待测血卡、唾液卡直接取样,操作简单,无需DNA的提取,2 h即可完成检测分析,通过对95份直扩样本的分型检测,验证了体系的直接扩增能力以及对三大洲际人群族群推断的准确性.除1份唾液卡样本出现大量等位基因丢失未获得有效分型,其余94份样本均获得有效分型,其原因可能是该唾液样本含有某些PCR抑制物[16 ] .同时,相较于微测序-CE技术检测SNP,基于直接PCR-CE技术的InDel分型方法对混合样本的检测更具优势,可以将混合样本中不同底物浓度反应至峰高值上,通过检测峰高值的不均衡来判别DNA样本是否为混合样本[6 -8 ] ,如SANTOS等[17 ] 对46-plex InDel体系进行实验室间比对,所有参与单位均能判断出待测样本为混合样本[18 ] 在35个InDel位点荧光复合扩增体系的构建与验证中,增大了主要贡献与次要贡献的混合比例,将两者比例设置为1∶19,当比例达到1∶19时,得到的峰型图与单样本时一致,即次要贡献者成分无法显示.后续将进一步增大混合比例,观察主要贡献者与次要贡献者的分型情况. ...
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...
A fully integrated and automated microsystem for rapid pharmacogenetic typing of multiple warfarin-related single-nucleotide polymorphisms
1
2016
... 根据SWGDAM指南,验证了本体系的准确性、案件样本的适用性以及非人源DNA样本的排除能力,并对体系的灵敏度及抗抑制剂能力进行了测试,反应体系的最低检测限为0.062 5 ng;当反应体系中腐殖酸质量浓度高于100 ng/μL时,部分等位基因出现丢失,与解通[18 ] 对35个InDel位点的研究结果相一致.与同样可区分三大洲际人群的SNP体系相比,本体系优势为基于PCR-CE技术,无需额外购置仪器,检测时间短.与其他InDel族群推断体系相比[7 ,10 ] ,本体系使用国产试剂,检测成本低,便于推广,可直接扩增,操作简单,同时,实验已证明可将该体系集成于微流控芯片系统[11 ] ,利用芯片检控仪自动完成对口腔拭子、血卡等样本的全集成检测,简化了操作步骤,降低了污染风险[19 ] ,实现“样本进-结果出”的一体化InDel检测. ...