4种深度学习图像分类算法在人工智能硅藻检验中的比较
朱永正 , 1 , 2 , * , 张吉 , 2 , * , 程奇 2 , 3 , 于慧潇 2 , 4 , 邓恺飞 2 , 张建华 2 , 秦志强 2 , 赵建 5 , 孙俊红 , 1 , 黄平 , 1 , 2
1.山西医科大学法医学院,山西 太原 030001
2.司法鉴定科学研究院 上海市法医学重点实验室 司法部司法鉴定重点实验室 上海市司法鉴定专业技术服务平台,上海 200063
3.贵州医科大学法医学院,贵州 贵阳 550000
4.徐州医科大学法医学教研室,江苏 徐州 221004
5.广州市刑事科学技术研究所 法医病理学公安部重点实验室,广东 广州 510442
Comparison among Four Deep Learning Image Classification Algorithms in AI-based Diatom Test
ZHU Yong-zheng , 1 , 2 , * , ZHANG Ji , 2 , * , CHENG Qi 2 , 3 , YU Hui-xiao 2 , 4 , DENG Kai-fei 2 , ZHANG Jian-hua 2 , QIN Zhi-qiang 2 , ZHAO Jian 5 , SUN Jun-hong , 1 , HUANG Ping , 1 , 2
1.School of Forensic Medicine, Shanxi Medical University, Taiyuan 030001, China
2.Shanghai Key Laboratory of Forensic Medicine, Key Laboratory of Forensic Science, Ministry of Justice, Shanghai Forensic Service Platform, Academy of Forensic Science, Shanghai 200063, China
3.Department of Forensic Medicine, Guizhou Medical University, Guiyang 550000, China
4.Department of Forensic Medicine, Xuzhou Medical University, Xuzhou 221004, Jiangsu Province, China
5.Guangzhou Forensic Science Institute & Key Laboratory of Forensic Pathology, Ministry of Public Security, Guangzhou 510442, China
通讯作者: 黄平,男,博士,研究员,主任法医师,主要从事法医病理学研究;E-mail:huangp@ssfjd.cn 孙俊红,男,教授,博士研究生导师,主要从事损伤病理学和猝死病理学研究;E-mail:junhong.sun@sxmu.edu.cn
第一联系人: 朱永正和张吉为共同第一作者
编委: 李正东
收稿日期: 2021-10-04
基金资助:
国家自然科学基金资助项目 . 82072115 上海市法医学重点实验室资助项目 . 20DZ2270800 司法部司法鉴定重点实验室资助项目 上海市司法鉴定专业技术服务平台资助项目 . 19DZ2292700 中央级公益性科研院所资助项目 . GY2020G-2
摘要
目的 选择深度学习图像分类算法中复杂性和准确性较为平衡的4种算法进行硅藻的自动识别,探究最适用于硅藻识别的分类算法,为法医学自动化硅藻检验研究提供数据参考。 方法 建立真实水中尸体肺组织消化液涂片的“硅藻”“背景”小样本量数据集(20 000张),用于4种算法(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)模型的训练、验证和测试。绘制受试者工作特征曲线、混淆矩阵并计算召回率、查准率、特异性、准确率及F 1分数,对各模型性能进行系统性评估。 结果 InceptionV3的硅藻识别性能明显优于其他3种算法,具有更为均衡的硅藻查全(89.80%)与查准(92.58%)性能;VGG16和Inception-ResNet-V2的硅藻识别性能相当,虽无法做到硅藻查全与查准的性能均衡,但其识别能力尚可接受;ResNet50的硅藻识别性能最低,其召回率仅为55.35%。在特征提取上,4种模型均提取到了硅藻和背景的特征,且都以硅藻区域为主要识别依据。 结论 包含Inception结构的模型,在硅藻特征提取方面具有更强的指向性和靶向性。其中,InceptionV3算法能够更为准确、靶向地提取到硅藻特征,具有最优的硅藻识别性能,更适合应用于日常法医学硅藻检验。
关键词:
法医病理学 人工智能 溺死 深度学习 硅藻检验 卷积神经网络
Abstract
Objective To select four algorithms with relatively balanced complexity and accuracy among deep learning image classification algorithms for automatic diatom recognition, and to explore the most suitable classification algorithm for diatom recognition to provide data reference for automatic diatom testing research in forensic medicine. Methods The “diatom” and “background” small sample size data set (20 000 images) of digestive fluid smear of corpse lung tissue in water were built to train, validate and test four convolutional neural network (CNN) models, including VGG16, ResNet50, InceptionV3 and Inception-ResNet-V2. The receiver operating characteristic curve (ROC) of subjects and confusion matrixes were drawn, recall rate, precision rate, specificity, accuracy rate and F 1 score were calculated, and the performance of each model was systematically evaluated. Results The InceptionV3 model achieved much better results than the other three models with a balanced recall rate of 89.80%, a precision rate of 92.58%. The VGG16 and Inception-ResNet-V2 had similar diatom recognition performance. Although the performance of diatom recall and precision detection could not be balanced, the recognition ability was acceptable. ResNet50 had the lowest diatom recognition performance, with a recall rate of 55.35%. In terms of feature extraction, the four models all extracted the features of diatom and background and mainly focused on diatom region as the main identification basis. Conclusion Including the Inception-dependent model, which has stronger directivity and targeting in feature extraction of diatom. The InceptionV3 achieved the best performance on diatom identification and feature extraction compared to the other three models. The InceptionV3 is more suitable for daily forensic diatom examination.
Keywords:
forensic pathology artificial intelligence drowning deep learning diatom test convolutional neural network
本文引用格式
朱永正, 张吉, 程奇, 于慧潇, 邓恺飞, 张建华, 秦志强, 赵建, 孙俊红, 黄平. 4种深度学习图像分类算法在人工智能硅藻检验中的比较 . 法医学杂志 [J], 2022, 38(1): 31-39 DOI:10.12116/j.issn.1004-5619.2021.411001
ZHU Yong-zheng, ZHANG Ji, CHENG Qi, YU Hui-xiao, DENG Kai-fei, ZHANG Jian-hua, QIN Zhi-qiang, ZHAO Jian, SUN Jun-hong, HUANG Ping. Comparison among Four Deep Learning Image Classification Algorithms in AI-based Diatom Test . Journal of Forensic Medicine 10.12116/j.issn.1004-5619.2021.411001
溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] 。硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] 。因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] 。然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] 。
近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] 。在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确。在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析。目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] 。图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] 。
ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平。基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本。涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成。目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行。
DL模型是DiatomNet V1.0的核心模块,对于软件自动化识别硅藻的能力起着举足轻重的作用。为不断优化软件的硅藻识别能力,探究最适合的算法模型,本研究采用小样本量数据集(20 000张图片)对不同发展阶段中具有代表性的4种深度学习算法(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)进行对比研究,探讨各模型结构、特征提取对硅藻识别性能的影响并明确最优分类算法,旨在为国内外从事法医学自动化硅藻检验研究的学者们提供算法选择方面的数据参考。
1 材料与方法
1.1 组织消化液涂片制备
为了训练和评估各DL算法在真实溺死样本中的性能,本研究根据《人体组织器官中硅藻硝酸破机法检验》(GA/T 813—2008)[27 ] 采用硝酸破机法分别对9例溺死尸体肺组织进行硅藻提取并制作成组织消化液涂片。具体步骤如下:提取死者左肺上叶并用超纯水进行清洗,随后在肺边缘区域切取15 g组织。为避免取材过程中出现交叉污染,上述操作使用一次性耗材及经蒸馏水反复清洗后的器具。将提取的组织切碎置于强酸溶液内,待其消化至澄清时,取10 mL消化液高速离心10 min(离心半径12.7 cm,4 000 r/min),弃上清液,将沉淀液均匀涂布于载玻片上。每个案例制作3张涂片,共27张。经本研究质量控制后从中剔除4张不合规涂片。本研究经司法鉴定科学研究院伦理委员会审核批准,涉及的人体标本均已获得死者家属的知情同意。
1.2 数据集建立
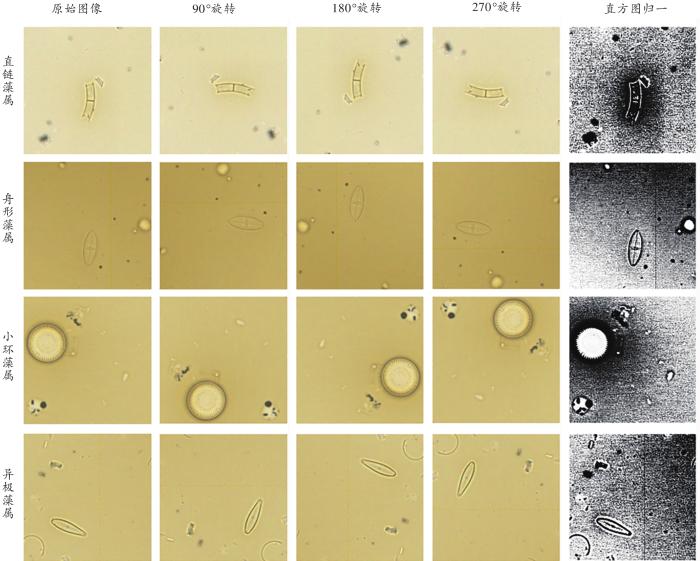
将尸体组织消化液涂片载入Aperio AT2高通量切片扫描仪(美国Leica公司)内进行数字化全切片扫描,扫描倍数为400,文件格式为svs(https://openslide.org/demo/ )。应用python 3.9.2软件以像素255×255对虚拟涂片进行分割。分割后的图片由2名经验丰富的法医病理学专家进行注释、分类。收集包含我国主要淡水硅藻种属(小环藻属、直链藻属、菱形藻属、舟形藻属、桥弯藻属、异极藻属等)的“硅藻”及“背景”样本,建立模型校正数据库。在23张涂片中,分别标记了11 364张硅藻和10 047张背景样本。为方便数据集的划分,数据库内保留“硅藻”和“背景”样本各10 000张。按6∶2∶2的比例将数据集分为训练集、验证集和测试集。为了保证模型的识别具有旋转和平移不变性,本研究对训练、验证集样本进行90°、180°、270°旋转和直方图归一化处理(图1 ),进一步扩充了数据库样本。根据损失函数,将训练集用于迭代校正模型参数,验证集则在每次迭代训练过程中用于动态评估CNN模型的性能,测试集则用于训练、优化后模型的独立性评估。测试集结果一般会低于训练和验证结果,但更接近于模型的真实效能及泛化能力。
图1
图1
数据增强后的模型训练数据集
Fig. 1
Model training data set after data enhancement
1.3 模型训练
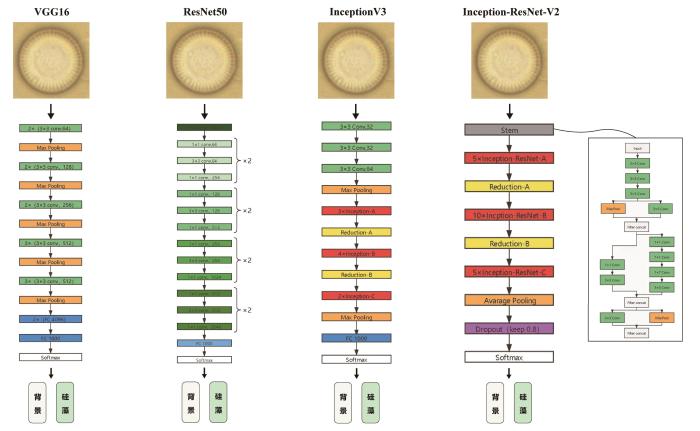
VGG16、ResNet50、InceptionV3、Inception-ResNet-V2 4种模型的架构详见图2 。为加速模型训练收敛及防止过拟合现象的出现,本研究使用迁移学习的方法对模型进行预训练,预训练数据集来自ImageNet数据库(https://image-net.org/index.php ),包含了1 000个目标分组的128万张图片[28 ] 。在训练过程中,主要参考验证集准确率和损失函数值。随着训练的迭代增加,模型逐渐趋于拟合,相应验证集准确率和损失函数值不再随之发生递增和递减。如出现相反情况,则提示有过拟合的情况发生。因此,在过拟合出现前,选取准确率最高或损失函数值最低的模型作为最优解。训练参数如下:学习率为0.001,单次训练图片64张,迭代次数上限设为150次,优化器为Adadelta。模型的训练在Ubuntu 18.04系统下完成,计算机工作站参数配置如下:CPU(Intel Xeon 6 132×2),GPU显卡(nVIDIA GeForce RTX2080Ti ×2),128 G内存。模型代码在python 3.6.5环境下运行,软件模块包括Tensorflow、OpenCV、Numpy、Pandas和Sklearn。
图2
图2
4种DL图像分类模型模式图
绿色表示卷积层;橙色表示池化层;蓝色表示全连接层;白色表示Softmax分类器;红色表示Inception模块。
Fig. 2
Flowchart of four DL image classification models
1.4 模型效能评价及比较
根据绘制受试者工作特征(receiver operating characteristic,ROC)曲线,计算其曲线下面积(area under curve,AUC)值。为了能够真实、全面地评估各项指标参数且不刻意优化单项指标结果[如召回率(recall rate,RR)或查准率(precision rate,PR)],本研究训练模型的ROC概率阈值固定为0.5。根据这一阈值统计模型在测试集预测中的真阳性(true positive,TP)数量、假阳性(false positive,FP)数量、真阴性(true negative,TN)数量、假阴性(false negative,FN)数量,并据此计算硅藻识别的RR、PR、F 1分数、特异性(specificity,S)及准确率(accuracy rate,AR)用于比较和评估4种模型架构的识别性能。各评估指标参数描述如下:
召回率也称为敏感性(sensitivity)、查全率,且查全率应用于硅藻检验的模型评估中更为切合,即识别正确的“硅藻”占样本中总硅藻的百分比,反映了硅藻识别的检出率,
R R = T P T P + F N (1)
查准率也称为精确率,且查准率应用于硅藻检验的模型评估中更为切合,主要是指真实硅藻在模型识别样本中的百分比,反映了硅藻识别的正确率,
P R = T P T P + F P (2)
特异性即识别正确的“背景”占样本中总背景的百分比,反映模型对背景样本的鉴别能力,
S = T N T N + F P (3)
准确率即所有正确识别的“硅藻”和“背景”占总样本的百分比,反映模型识别硅藻和背景的总体正确率,
A R = T P + T N T P + F N + T N + F P (4)
F 1分数:灵敏度和精确率的调和平均数,反映模型硅藻识别的综合性能,
F 1 = 2 × P R × R R P R + R R (5)
(1)~(5)公式中,TP 代表真阳性数量,即模型正确识别硅藻的样本数量;TN 代表真阴性数量,即模型正确识别背景的样本数量;FP 代表假阳性数量,即模型错误识别硅藻的样本数量;FN 代表假阴性数量,即模型错误识别背景的样本数量。
1.5 模型硅藻识别的可视化及可解释性分析
应用导向反向传播(guided-back propagation,GBP)算法和梯度加权类激活映射(gradient-weighted class activation mapping,Grad-CAM)算法对模型单个样本(中心纲及羽纹纲硅藻)预测能力进行可视化分析,明确模型所学习到的全部特征是否主要集中于硅藻区域。GBP可对卷积核本身进行可视化,能够“看到”模型较深卷积层所学习到的“硅藻”和“背景”的特征。Grad-CAM可通过热力图显示原始图片的不同区域对硅藻识别的“贡献”程度,明确在硅藻识别中起关键作用的特征信息[29 ] 。
2 结果
2.1 模型训练
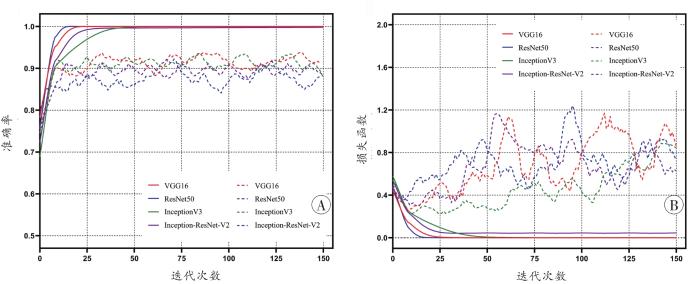
图3 及表1 反映了4种模型在训练过程中准确率和损失函数值随迭代次数增加而产生的变化。基于Inception架构的模型(包括InceptionV3和Inception-ResNet-V2)训练收敛速度明显快于其他两种模型。4种模型的训练集准确率均在99.50%以上。相较而言,InceptionV3模型的验证集准确率最高,Inception-ResNet-V2和VGG16模型次之,均在95.00%以上,ResNet50的准确率最低,仅为94.00%。
图3
图3
4种模型经二阶平滑处理的训练准确率及损失函数曲线
A:准确率曲线;B:损失函数曲线。实线表示训练集,虚线表示验证集。
Fig. 3
The accuracy and loss curves of four models after the 2nd order smoothing on training and validation
2.2 模型效能评价与比较
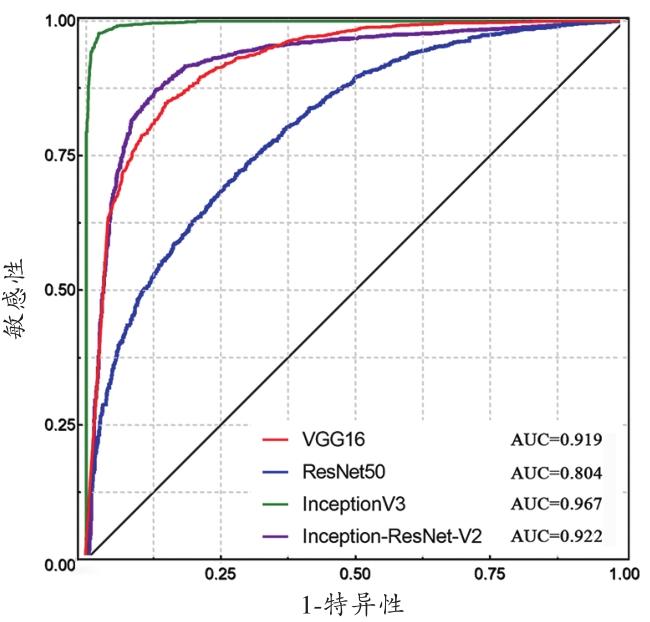
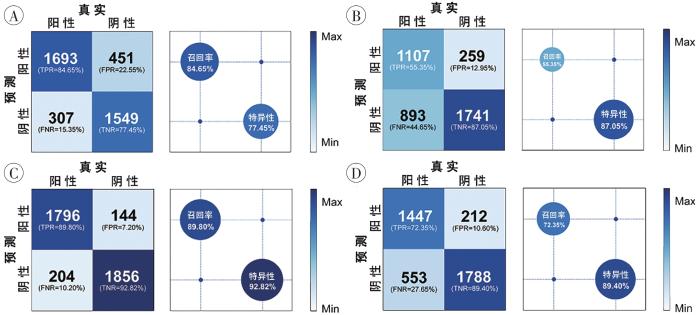
表2 和图4 ~5 显示,在模型测试过程中,InceptionV3模型的硅藻识别性能优于其他模型,其AUC(0.967)、准确率(91.30%)和特异性(92.80%)在4种模型中最高,且同时具有较高的召回率(89.80%)、查准率(92.58%)和F 1分数(0.91),提示该模型在区分硅藻和背景方面均具有较强的鉴识能力。VGG16和Inception-ResNet-V2模型整体性能不如InceptionV3,但仍具备准确识别硅藻的能力,其AUC值均在0.900以上。其中,VGG16具有相对较高的硅藻筛选能力(召回率为84.65%),而Inception-ResNet-V2硅藻识别精准率则相对较高(查准率为87.22%)。相较于InceptionV3,上述2个模型尚无法做到硅藻与背景鉴别能力的均衡。ResNet50模型各项评估指标最低。虽然其特异性(87.05%)和查准率(81.04%)尚可,但较低的召回率(55.35%)意味着几乎一半的硅藻会被漏检。
图4
图4
4种最优模型的ROC曲线及其AUC值
Fig. 4
The ROC curves and AUC values of 4 optimal models
图5
图5
4种最优模型的混淆矩阵、召回率和特异性
A:VGG16;B:ResNet50;C:InceptionV3;D:Inception-ResNet-V2。TPR表示真阳性率(true positive rate),FPR表示假阳性率(false positive rate),TNR表示真阴性率(true negative rate),FNR表示假阴性率(false negative rate)。
Fig. 5
The confusion matrixes, recall rate and specificity of 4 optimal models
2.3 模型硅藻识别可视化分析
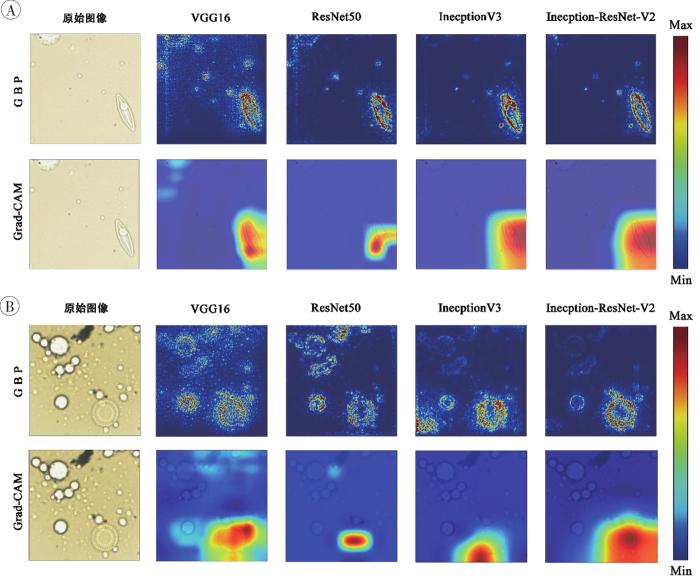
如图6 所示,应用Grad-CAM和GBP算法分别检验4种模型对中心纲和羽纹纲硅藻的聚焦程度,4种模型均在硅藻区域内展现了较高的关注强度,提示其均通过训练准确提取到了硅藻的形态学特征,但GBP结果反映InceptionV3提取到的硅藻特征更多且集中,更有利于其准确分类。
图6
图6
应用Grad-CAM和GBP方法对4种模型在中心纲及羽纹纲硅藻识别上的热图分析
A:中心纲分析结果;B:羽纹纲分析结果。红色像素代表模型高度关注的区域。
Fig. 6
Heat map analysis of pennataes and centricaes identificated with 4 models by Grad-CAM and GBP methods
3 讨论
近年来,深度学习技术发展迅速,已被广泛应用于医学图像识别[13 -14 ] 。在法医学硅藻检验领域,已有学者利用CNN中的目标检测算法和图像分类算法实现了硅藻的自动化检验,较传统人工检验方法更高效、准确[15 ] 。然而,目前的研究主要集中于单一算法在自动化硅藻检验上的实现和应用,缺乏各算法间的系统性比较研究。本研究选择了CNN图像分类算法中复杂性和准确性较为均衡的4种模型(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)进行硅藻的自动识别,并基于小样本数据量进行模型的训练、评估,探究他们在硅藻识别上的适用性,为国内外从事法医学自动化硅藻检验研究的学者提供一定的数据参考。
在众多模型评估指标中,召回率和查准率主要决定着模型是否能够真正应用于实践。较低的召回率会造成大量硅藻的漏检;而低查准率则会导致大量无效备选图片的生成,扫描半径越大,其对人工确认的负担也就越大,试想从数百、数千甚至数万张图片中(尤其是肝、肾组织)挑选少量硅藻,随之所产生的工作量无异于人工识别。因此,为了能够兼顾召回率和查准率2项指标,往往需要额外计算F 1分数值用于综合判断模型的实际预测能力。
经过对最优模型的选择和外部测试,4种模型对硅藻均具有一定的识别能力,且以硅藻区域为主要识别依据(图6 ),但InceptionV3模型明显优于其他模型,较高的F 1分数值提示该模型具有更为全面的识别性能,这主要得益于其独特的Inception结构,可将一个n ×n 的卷积核拆解成n ×1和1×n 个,在保持高性能计算的同时增加了网络的宽度和深度,可让模型灵活决定卷积核的大小,在一定程度上降低了训练参数的规模,从而有效提升了运算速度[30 ] 。此外,InceptionV3深且简单的模型架构,使其不易产生过拟合和梯度弥散等问题,能够更好、更准确地对目标物特征进行抽象化提取。
ResNet50是这4种模型中性能最差的,其召回率仅为55.35%,对法医学硅藻检验来说,意味着有一半的硅藻将会被漏检。此外,GBP热图分析发现,该模型提取到的特征较为分散,特征数量也相对较少。ResNet模型架构主要是基于残差学习的思想,可在训练过程中将部分参数信息以跳跃性的方式进行传输,在一定程度上保护了参数信息的完整性,省去了中间繁琐的参数调整过程,极大地简化了学习目标和难度系数[31 ] 。然而,可能正是这种残差学习方式,使得该模型没有学习到硅藻的关键形态学特征,间接导致其不能对涂片中的硅藻进行准确识别。
Inception-ResNet-V2和VGG16模型具有较好的硅藻识别性能,在GBP热图分析中,Inception-ResNet-V2比VGG16提取到的特征更靶向集中于硅藻,且硅藻的分类特征更多,但二者在硅藻识别上都无法兼顾硅藻查全和查准性能。VGG16对硅藻识别的特异性较低,会有很多的“背景”被误检;而Inception-ResNet-V2模型对硅藻识别的召回率较低,会有很多的“硅藻”被漏检。从模型本身因素考虑,VGG16是这4种算法中结构最为简单的模型,常用于图像特征的提取、目标检测候选框的生成,但其最大的问题在于训练参数过多,约有1.38亿个,更容易产生过拟合现象[21 ] 。由于VGG模型具有搭建简单、硬件要求低及性能适中的优点,因此,其更合符合小批量、简单样本的训练;Inception-ResNet-V2是在Inception结构的基础上引入了残差网络的思想,将浅层特征通过另外一条分支累加至更为高层的特征结构中,使得部分特征信息得以重复利用,有效避免了深层网络梯度弥散的问题[22 ] 。与ResNet50相似的是,该模型仍有可能会漏掉部分必要形态学特征,但自身存在的Inception结构使其继承了InceptionV3部分特征提取的优势,因此,Inception-ResNet-V2模型整体性能介于InceptionV3和ResNet50之间。
在法医学自动化硅藻检验中,主要用到以CNN为核心的图片分类算法和目标检测算法。本研究结果发现,InceptionV3的各项评估指标(如查准率、召回率、F 1分数等)几乎均达到了90%或以上,如进行更大样本量的训练(如DiatomNet V1.0中的模型)其效能将会得到进一步提升。就本研究结果而言,InceptionV3模型的效能(F 1分数0.91,查准率92.58%,召回率89.89%,概率阈值0.5)均高于已发表的目标检测算法,如KRAUSE等[32 ] 的自建模型(F 1分数0.82,查准率77%,召回率87%)、RUIZ-SANTAQUITERIA等[33 ] 的Mask R-CNN(F 1分数0.85,查准率85%,召回率86%)、BUENO等[34 ] 的YOLO(F 1分数0.78,查准率75%,召回率84%)。此外,本研究所采用的数字化WSI扫描技术能够在数分钟内获取整张涂片的数据,结合InceptionV3的高性能分类能力,使得DiatomNet V1.0无论在时间消耗、检测准确率、人工确认负担等方面均具有突出的优势,具有一定的实际应用前景。然而,不可否认的是,该系统主要基于传统光镜识别,放大倍数仅400倍,难以对硅藻种属进行准确判断。尽管ZHANG等[24 ] 的动物实验证实基于InceptionV3模型的硅藻种属分类方法能够大致推断出位于上海市黄浦江、苏州河水域的溺水地点,但为了获取更为精准的预测结果,仍需要基因测序、扫描电子显微镜等技术手段对硅藻种属进行更为细致的分类。
综上,通过对4种模型的比较,InceptionV3具有最优的硅藻识别性能且能够准确、靶向提取到硅藻特征,更适合应用于法医学实践。虽然Inception-ResNet-V2模型的硅藻识别性能一般,但其特征的提取也是集中于硅藻本身。由此得出,具有Inception结构的模型更适合硅藻识别任务。
参考文献
View Option
[1]
World Health Organization Global report on drowning: Preventing a leading killer
[R/OL]. (2014-09-17 )[2021-09-21 ]. .
[本文引用: 1]
[3]
[本文引用: 1]
LI Q MA K J ZHANG X D et al Diatom test in lung tissue of corpses in water and causes of death
[J]. Fayixue Zazhi ,2011 ,27 (5 ):324 -326 ,333 .
[本文引用: 1]
[4]
CARBALLEIRA R VIEIRA D N FEBRERO-BANDE M et al A valid method to determine the site of drowning
[J]. Int J Legal Med ,2018 ,132 (2 ):487 -497 . doi:10.1007/s00414-017-1708-1 .
[6]
LUNETTA P PENTTILÄ A HÄLLFORS G Scanning and transmission electron microscopical evidence of the capacity of diatoms to penetrate the alveolo-capillary barrier in drowning
[J]. Int J Legal Med ,1998 ,111 (5 ):229 -237 . doi:10.1007/s004140050159 .
[本文引用: 1]
[7]
ZHANG P KANG X ZHANG S et al The length and width of diatoms in drowning cases as the evidence of diatoms penetrating the alveoli-capillary barrier
[J]. Int J Legal Med ,2020 ,134 (3 ):1037 -1042 . doi:10.1007/s00414-019-02164-4 .
[本文引用: 1]
[8]
LUDES B COSTE M NORTH N et al Diatom analysis in victim’s tissues as an indicator of the site of drowning
[J]. Int J Legal Med ,1999 ,112 (3 ):163 -166 . doi:10.1007/s004140050224 .
[本文引用: 1]
[9]
SHEN X LIU Y XIAO C et al Analysis of false-positive results of diatom test in the diagnosis of drowning-would not be an impediment
[J]. Int J Legal Med ,2019 ,133 (6 ):1819 -1824 . doi:10.1007/s00414-019-02021-4 .
[本文引用: 1]
[10]
BORTOLOTTI F TAGLIARO F MANETTO G Objective diagnosis of drowning by the “diatom test” - A critical review
[J]. Forensic Sci Rev ,2004 ,16 (2 ):135 -148 .
[本文引用: 1]
[11]
[本文引用: 1]
ZHOU Y Y CAO Y J YANG Y et al Application of artificial intelligence automatic diatom identification system in practical cases
[J]. Fayixue Zazhi ,2020 ,36 (2 ):239 -242 .
[本文引用: 1]
[12]
KRIZHEVSKY A SUTSKEVER I HINTON G E ImageNet classification with deep convolutional neural networks
[J]. Commun ACM ,2017 ,60 (6 ):84 -90 . doi:10.1145/3065386 .
[本文引用: 2]
[14]
HA Q S LIU B LIU F X Identifying melanoma images using EfficientNet ensemble: Winning solution to the SIIM-ISIC melanoma classification challenge
[J/OL]. (2020-10-11 )[2021-09-21 ]. arXiv:2010 . 05351 v1 .
[本文引用: 2]
[16]
HU H GU J Y ZHANG Z et al Relation networks for object detection
[C]//2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition . Salt Lake City :IEEE ,2018 :3588 -3597 . doi:10.1109/CVPR.2018.00378 .
[本文引用: 1]
[17]
GIRSHICK R DONAHUE J DARRELL T et al Rich feature hierarchies for accurate object detection and semantic segmentation
[C]//2014 IEEE Conference on Computer Vision and Pattern Recognition . Columbus :IEEE ,2014 :580 -587 . doi:10.1109/CVPR.2014.81 .
[18]
GIRSHICK R Fast R-CNN
[C]//2015 IEEE International Conference on Computer Vision . Santiago :IEEE ,2015 :1440 -1448 . doi:10.1109/ICCV. 2015.169 .
[19]
REN S Q HE K M GIRSHICK R et al Faster R-CNN: Towards real-time object detection with region proposal networks
[J]. IEEE Trans Pattern Anal Mach Intell,2017 ,39 (6 ):1137 -1149 . doi:10.1109/TPAMI.2016.2577031 .
[20]
YU W M XUE Y KNOOPS R et al Automated diatom searching in the digital scanning electron microscopy images of drowning cases using the deep neural networks
[J]. Int J Leg Med ,2021 ,135 (2 ):497 -508 . doi:10.1007/s00414-020-02392-z .
[本文引用: 1]
[21]
SIMONYAN K ZISSERMAN A Very deep convolutional networks for large-scale image recognition
[J/OL].(2015-04-10 )[2021-09-21 ]. arXiv:.
[本文引用: 2]
[22]
SZEGEDY C IOFFE S VANHOUCKE V Inception-V
4 , inception-ResNet and the impact of residual connections on learning[J/OL]. (2016-08-23 )[2021-09-21 ]. arXiv:.
[本文引用: 2]
[23]
ZHOU Y Y ZHANG J HUANG J et al Digital whole-slide image analysis for automated diatom test in forensic cases of drowning using a convolutional neural network algorithm
[J]. Forensic Sci Int ,2019 ,302 :109922 . doi:10.1016/j.forsciint.2019. 109922 .
[本文引用: 1]
[24]
ZHANG J ZHOU Y VIEIRA D N et al An efficient method for building a database of diatom populations for drowning site inference using a deep learning algorithm
[J]. Int J Legal Med ,2021 ,135 (3 ):817 -827 . doi:10.1007/s00414-020-02497-5 .
[本文引用: 2]
[25]
黄平 ,张吉 ,邓恺飞 ,等 一种基于硅藻种属分类的溺水地点鉴定系统及方法
:1102916.7 [P]. 2021-07-20 .
[本文引用: 1]
HUANG P ZHANG J DENG K F et al A drowning site identification system and method based on the classification of diatom species
:1102916.7 [P]. 2021-07-02 .
[本文引用: 1]
[26]
司法鉴定科学研究院 智能化硅藻鉴识系统
[简称 :DiatomNet ]V1.0:2021SR0337617 [P]. 2020-12-23 .
[本文引用: 1]
Accademy of Forensic Science Intelligent diatom discrimination system
[Abbreviation : DiatomNet ]V1.0:2021SR0337617 [P]. 2020-12-23 .
[本文引用: 1]
[27]
福建省公安厅刑侦总队 人体组织器官中硅藻硝酸破机法检验 : [S]. 北京 :中国标准出版社 ,2009 .
[本文引用: 1]
Criminal Investigation Corps of Fujian Provincial Public Security Department Detection the diatoms in human tissues and organs by digesting organic matter with nitric acid : [S]. Beijing : China Standards Publishing Press ,2009 .
[本文引用: 1]
[28]
DENG J DONG W SOCHER R et al ImageNet: A large-scale hierarchical image database
[C]//2009 IEEE Conference on Computer Vision and Pattern Recognition .Miami : IEEE ,2009 :248 -255 . doi:10.1109/CVPR.2009.5206848 .
[本文引用: 1]
[29]
SELVARAJU R R COGSWELL M DAS A , et al Grad-CAM: Visual explanations from deep networks via gradient-based localization
[J]. 2017 IEEE Int Conf Comput Vis ICCV ,2017 :618 -626 . doi:10.1109/ICCV.2017.74 .
[本文引用: 1]
[30]
SZEGEDY C VANHOUCKE V IOFFE S et al Rethinking the inception architecture for computer vision
[J]. 2016 IEEE Conf Comput Vis Pattern Recognit CVPR ,2016 :2818 -2826 . doi:10.1109/CVPR.2016.308 .
[本文引用: 1]
[31]
HE K M ZHANG X Y REN S Q et al Deep residual learning for image recognition
[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition . Las Vegas : IEEE ,2016 :770 -778 . doi:10.1109/CVPR.2016.90 .
[本文引用: 1]
[32]
KRAUSE L M K KOC J , ROSENHAHN B et al Fully convolutional neural network for detection and counting of diatoms on coatings after short-term field exposure
[J]. Environ Sci Technol ,2020 ,54 (16 ):10022 -10030 . doi:10.1021/acs.est.0c01982 .
[本文引用: 1]
[33]
RUIZ-SANTAQUITERIA J BUENO G DENIZ O et al Semantic versus instance segmentation in microscopic algae detection
[J]. Eng Appl Artif Intel ,2020 ,87 . doi:10.1016/j.engappai.2019.103271 .
[本文引用: 1]
[34]
BUENO G DENIZ O RUIZ-SANTAQUITERIA J et al Lights and pitfalls of convolutional neural networks for diatom identification
[C]//Optics, Photonics and Digital Technologies for Imaging Applications V . Strasbourg :SPIE Photonics Europe ,2018 . doi:10.1117/12.2309488 .
[本文引用: 1]
Global report on drowning: Preventing a leading killer
1
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
Management for the drowning patient
1
2021
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
水中尸体肺组织硅藻检验与死因分析
1
2011
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
水中尸体肺组织硅藻检验与死因分析
1
2011
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
A valid method to determine the site of drowning
0
2018
Drowning: Still a difficult autopsy diagnosis
2
2006
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
... [5 ,8 -9 ].然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
Scanning and transmission electron microscopical evidence of the capacity of diatoms to penetrate the alveolo-capillary barrier in drowning
1
1998
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
The length and width of diatoms in drowning cases as the evidence of diatoms penetrating the alveoli-capillary barrier
1
2020
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
Diatom analysis in victim’s tissues as an indicator of the site of drowning
1
1999
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
Analysis of false-positive results of diatom test in the diagnosis of drowning-would not be an impediment
1
2019
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
Objective diagnosis of drowning by the “diatom test” - A critical review
1
2004
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
基于人工智能硅藻自动化识别系统的实际案例应用
1
2020
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
基于人工智能硅藻自动化识别系统的实际案例应用
1
2020
... 溺死是世界范围内第三大意外死亡原因[1 -2 ] ,在法医学实践中,由于受到尸体腐败和水中生物破坏的影响,溺死的诊断极具挑战性[3 -5 ] .硅藻,一种广泛栖息于水生环境的浮游生物,会在溺水过程中随着溺液被吸入肺组织,并通过肺泡壁-毛细血管屏障进入血液循环,分布到肝、肾等其他实质器官[6 -7 ] .因此,硅藻检验被视为辅助溺死诊断的“金标准”,是主要的实验室检验方法[5 ,8 -9 ] .然而传统硅藻检验方法需要专业人员利用光镜或电子显微镜对硅藻进行观察和计数,耗费了大量时间和精力,且检验结果具有一定的主观性[10 -11 ] . ...
ImageNet classification with deep convolutional neural networks
2
2017
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... [12 ,21 -22 ]. ...
BACH: Grand challenge on breast cancer histology images
2
2019
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... 近年来,深度学习技术发展迅速,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者利用CNN中的目标检测算法和图像分类算法实现了硅藻的自动化检验,较传统人工检验方法更高效、准确[15 ] .然而,目前的研究主要集中于单一算法在自动化硅藻检验上的实现和应用,缺乏各算法间的系统性比较研究.本研究选择了CNN图像分类算法中复杂性和准确性较为均衡的4种模型(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)进行硅藻的自动识别,并基于小样本数据量进行模型的训练、评估,探究他们在硅藻识别上的适用性,为国内外从事法医学自动化硅藻检验研究的学者提供一定的数据参考. ...
Identifying melanoma images using EfficientNet ensemble: Winning solution to the SIIM-ISIC melanoma classification challenge
2
2010
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... 近年来,深度学习技术发展迅速,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者利用CNN中的目标检测算法和图像分类算法实现了硅藻的自动化检验,较传统人工检验方法更高效、准确[15 ] .然而,目前的研究主要集中于单一算法在自动化硅藻检验上的实现和应用,缺乏各算法间的系统性比较研究.本研究选择了CNN图像分类算法中复杂性和准确性较为均衡的4种模型(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)进行硅藻的自动识别,并基于小样本数据量进行模型的训练、评估,探究他们在硅藻识别上的适用性,为国内外从事法医学自动化硅藻检验研究的学者提供一定的数据参考. ...
Research advances in forensic diatom testing
2
2020
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... 近年来,深度学习技术发展迅速,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者利用CNN中的目标检测算法和图像分类算法实现了硅藻的自动化检验,较传统人工检验方法更高效、准确[15 ] .然而,目前的研究主要集中于单一算法在自动化硅藻检验上的实现和应用,缺乏各算法间的系统性比较研究.本研究选择了CNN图像分类算法中复杂性和准确性较为均衡的4种模型(VGG16、ResNet50、InceptionV3和Inception-ResNet-V2)进行硅藻的自动识别,并基于小样本数据量进行模型的训练、评估,探究他们在硅藻识别上的适用性,为国内外从事法医学自动化硅藻检验研究的学者提供一定的数据参考. ...
Relation networks for object detection
1
2018
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
Rich feature hierarchies for accurate object detection and semantic segmentation
0
2014
Faster R-CNN: Towards real-time object detection with region proposal networks
0
2017
Automated diatom searching in the digital scanning electron microscopy images of drowning cases using the deep neural networks
1
2021
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
Very deep convolutional networks for large-scale image recognition
2
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... Inception-ResNet-V2和VGG16模型具有较好的硅藻识别性能,在GBP热图分析中,Inception-ResNet-V2比VGG16提取到的特征更靶向集中于硅藻,且硅藻的分类特征更多,但二者在硅藻识别上都无法兼顾硅藻查全和查准性能.VGG16对硅藻识别的特异性较低,会有很多的“背景”被误检;而Inception-ResNet-V2模型对硅藻识别的召回率较低,会有很多的“硅藻”被漏检.从模型本身因素考虑,VGG16是这4种算法中结构最为简单的模型,常用于图像特征的提取、目标检测候选框的生成,但其最大的问题在于训练参数过多,约有1.38亿个,更容易产生过拟合现象[21 ] .由于VGG模型具有搭建简单、硬件要求低及性能适中的优点,因此,其更合符合小批量、简单样本的训练;Inception-ResNet-V2是在Inception结构的基础上引入了残差网络的思想,将浅层特征通过另外一条分支累加至更为高层的特征结构中,使得部分特征信息得以重复利用,有效避免了深层网络梯度弥散的问题[22 ] .与ResNet50相似的是,该模型仍有可能会漏掉部分必要形态学特征,但自身存在的Inception结构使其继承了InceptionV3部分特征提取的优势,因此,Inception-ResNet-V2模型整体性能介于InceptionV3和ResNet50之间. ...
Inception-V
2
4
... 近年来,有关人工智能(artificial intelligence,AI)领域中深度学习(deep learning,DL)的研究发展迅速,在目标检测、图像分类等任务上取得了不错的成绩[12 ] ,已被广泛应用于医学图像识别[13 -14 ] .在法医学硅藻检验领域,已有学者[15 ] 利用DL实现了硅藻的自动化检验,使检验更加高效、准确.在目前的研究中,主要应用DL中卷积神经网络(convolutional neural network,CNN)两大算法,即目标检测算法和图像分类算法,来实现涂片背景中硅藻的自动化分析.目标检测算法通过获取背景中硅藻的候选框进行识别和分类,代表算法有R-CNN、Fast R-CNN、Faster R-CNN、RetinaNet等[16 -20 ] .图像分类算法是将图片中的“硅藻”和“背景”进行分类标注,通过输出图片的分类标签概率来实现硅藻的识别和分类,代表算法有AlexNet、VGG、ResNet、Inception和Inception-ResNet等[12 ,21 -22 ] . ...
... Inception-ResNet-V2和VGG16模型具有较好的硅藻识别性能,在GBP热图分析中,Inception-ResNet-V2比VGG16提取到的特征更靶向集中于硅藻,且硅藻的分类特征更多,但二者在硅藻识别上都无法兼顾硅藻查全和查准性能.VGG16对硅藻识别的特异性较低,会有很多的“背景”被误检;而Inception-ResNet-V2模型对硅藻识别的召回率较低,会有很多的“硅藻”被漏检.从模型本身因素考虑,VGG16是这4种算法中结构最为简单的模型,常用于图像特征的提取、目标检测候选框的生成,但其最大的问题在于训练参数过多,约有1.38亿个,更容易产生过拟合现象[21 ] .由于VGG模型具有搭建简单、硬件要求低及性能适中的优点,因此,其更合符合小批量、简单样本的训练;Inception-ResNet-V2是在Inception结构的基础上引入了残差网络的思想,将浅层特征通过另外一条分支累加至更为高层的特征结构中,使得部分特征信息得以重复利用,有效避免了深层网络梯度弥散的问题[22 ] .与ResNet50相似的是,该模型仍有可能会漏掉部分必要形态学特征,但自身存在的Inception结构使其继承了InceptionV3部分特征提取的优势,因此,Inception-ResNet-V2模型整体性能介于InceptionV3和ResNet50之间. ...
Digital whole-slide image analysis for automated diatom test in forensic cases of drowning using a convolutional neural network algorithm
1
2019
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
An efficient method for building a database of diatom populations for drowning site inference using a deep learning algorithm
2
2021
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
... 在法医学自动化硅藻检验中,主要用到以CNN为核心的图片分类算法和目标检测算法.本研究结果发现,InceptionV3的各项评估指标(如查准率、召回率、F 1分数等)几乎均达到了90%或以上,如进行更大样本量的训练(如DiatomNet V1.0中的模型)其效能将会得到进一步提升.就本研究结果而言,InceptionV3模型的效能(F 1分数0.91,查准率92.58%,召回率89.89%,概率阈值0.5)均高于已发表的目标检测算法,如KRAUSE等[32 ] 的自建模型(F 1分数0.82,查准率77%,召回率87%)、RUIZ-SANTAQUITERIA等[33 ] 的Mask R-CNN(F 1分数0.85,查准率85%,召回率86%)、BUENO等[34 ] 的YOLO(F 1分数0.78,查准率75%,召回率84%).此外,本研究所采用的数字化WSI扫描技术能够在数分钟内获取整张涂片的数据,结合InceptionV3的高性能分类能力,使得DiatomNet V1.0无论在时间消耗、检测准确率、人工确认负担等方面均具有突出的优势,具有一定的实际应用前景.然而,不可否认的是,该系统主要基于传统光镜识别,放大倍数仅400倍,难以对硅藻种属进行准确判断.尽管ZHANG等[24 ] 的动物实验证实基于InceptionV3模型的硅藻种属分类方法能够大致推断出位于上海市黄浦江、苏州河水域的溺水地点,但为了获取更为精准的预测结果,仍需要基因测序、扫描电子显微镜等技术手段对硅藻种属进行更为细致的分类. ...
一种基于硅藻种属分类的溺水地点鉴定系统及方法
1
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
一种基于硅藻种属分类的溺水地点鉴定系统及方法
1
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
智能化硅藻鉴识系统
1
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
智能化硅藻鉴识系统
1
... ZHOU等[23 -24 ] 先后将DL技术应用于法医学硅藻检验,即通过InceptionV3算法、数字化全切片图像(whole slide image,WSI)技术和强酸消化法的联用对组织消化液涂片中的硅藻进行自动化识别和分类,其识别准确率及种属分类准确率分别已达到甚至超过法医病理学家人工识别诊断的水平.基于上述研究成果,本团队现已获授权相关国家发明专利(授权号CN 110851632 B)[25 ] 并独立开发出一套智能化硅藻鉴识软件系统,即DiatomNet V1.0[26 ] (软件著作权登记号2021SR0337617),其能够在400倍光镜下自动识别标准涂片(半径大于1.5 cm)中的所有硅藻及其碎片,大幅度降低了人工识别所带来的时间和精力成本.涉及涂片扫描(所需时间10 min以内)、数字化涂片预处理(所需时间8 min左右)、自动化硅藻识别(所需时间5~6 min)、硅藻检验结果呈现、人工结果确认(所需时间约6 min)、检测报告生成在内的全底物硅藻检验仅需要30~40 min,最快可在25 min左右完成.目前,该软件已在司法鉴定科学研究院法医病理学研究室进行测试,相关程序代码优化工作还在稳步进行. ...
1
2009
... 为了训练和评估各DL算法在真实溺死样本中的性能,本研究根据《人体组织器官中硅藻硝酸破机法检验》(GA/T 813—2008)[27 ] 采用硝酸破机法分别对9例溺死尸体肺组织进行硅藻提取并制作成组织消化液涂片.具体步骤如下:提取死者左肺上叶并用超纯水进行清洗,随后在肺边缘区域切取15 g组织.为避免取材过程中出现交叉污染,上述操作使用一次性耗材及经蒸馏水反复清洗后的器具.将提取的组织切碎置于强酸溶液内,待其消化至澄清时,取10 mL消化液高速离心10 min(离心半径12.7 cm,4 000 r/min),弃上清液,将沉淀液均匀涂布于载玻片上.每个案例制作3张涂片,共27张.经本研究质量控制后从中剔除4张不合规涂片.本研究经司法鉴定科学研究院伦理委员会审核批准,涉及的人体标本均已获得死者家属的知情同意. ...
1
2009
... 为了训练和评估各DL算法在真实溺死样本中的性能,本研究根据《人体组织器官中硅藻硝酸破机法检验》(GA/T 813—2008)[27 ] 采用硝酸破机法分别对9例溺死尸体肺组织进行硅藻提取并制作成组织消化液涂片.具体步骤如下:提取死者左肺上叶并用超纯水进行清洗,随后在肺边缘区域切取15 g组织.为避免取材过程中出现交叉污染,上述操作使用一次性耗材及经蒸馏水反复清洗后的器具.将提取的组织切碎置于强酸溶液内,待其消化至澄清时,取10 mL消化液高速离心10 min(离心半径12.7 cm,4 000 r/min),弃上清液,将沉淀液均匀涂布于载玻片上.每个案例制作3张涂片,共27张.经本研究质量控制后从中剔除4张不合规涂片.本研究经司法鉴定科学研究院伦理委员会审核批准,涉及的人体标本均已获得死者家属的知情同意. ...
ImageNet: A large-scale hierarchical image database
1
2009
... VGG16、ResNet50、InceptionV3、Inception-ResNet-V2 4种模型的架构详见图2 .为加速模型训练收敛及防止过拟合现象的出现,本研究使用迁移学习的方法对模型进行预训练,预训练数据集来自ImageNet数据库(https://image-net.org/index.php ),包含了1 000个目标分组的128万张图片[28 ] .在训练过程中,主要参考验证集准确率和损失函数值.随着训练的迭代增加,模型逐渐趋于拟合,相应验证集准确率和损失函数值不再随之发生递增和递减.如出现相反情况,则提示有过拟合的情况发生.因此,在过拟合出现前,选取准确率最高或损失函数值最低的模型作为最优解.训练参数如下:学习率为0.001,单次训练图片64张,迭代次数上限设为150次,优化器为Adadelta.模型的训练在Ubuntu 18.04系统下完成,计算机工作站参数配置如下:CPU(Intel Xeon 6 132×2),GPU显卡(nVIDIA GeForce RTX2080Ti ×2),128 G内存.模型代码在python 3.6.5环境下运行,软件模块包括Tensorflow、OpenCV、Numpy、Pandas和Sklearn. ...
Grad-CAM: Visual explanations from deep networks via gradient-based localization
1
2017
... 应用导向反向传播(guided-back propagation,GBP)算法和梯度加权类激活映射(gradient-weighted class activation mapping,Grad-CAM)算法对模型单个样本(中心纲及羽纹纲硅藻)预测能力进行可视化分析,明确模型所学习到的全部特征是否主要集中于硅藻区域.GBP可对卷积核本身进行可视化,能够“看到”模型较深卷积层所学习到的“硅藻”和“背景”的特征.Grad-CAM可通过热力图显示原始图片的不同区域对硅藻识别的“贡献”程度,明确在硅藻识别中起关键作用的特征信息[29 ] . ...
Rethinking the inception architecture for computer vision
1
2016
... 经过对最优模型的选择和外部测试,4种模型对硅藻均具有一定的识别能力,且以硅藻区域为主要识别依据(图6 ),但InceptionV3模型明显优于其他模型,较高的F 1分数值提示该模型具有更为全面的识别性能,这主要得益于其独特的Inception结构,可将一个n ×n 的卷积核拆解成n ×1和1×n 个,在保持高性能计算的同时增加了网络的宽度和深度,可让模型灵活决定卷积核的大小,在一定程度上降低了训练参数的规模,从而有效提升了运算速度[30 ] .此外,InceptionV3深且简单的模型架构,使其不易产生过拟合和梯度弥散等问题,能够更好、更准确地对目标物特征进行抽象化提取. ...
Deep residual learning for image recognition
1
2016
... ResNet50是这4种模型中性能最差的,其召回率仅为55.35%,对法医学硅藻检验来说,意味着有一半的硅藻将会被漏检.此外,GBP热图分析发现,该模型提取到的特征较为分散,特征数量也相对较少.ResNet模型架构主要是基于残差学习的思想,可在训练过程中将部分参数信息以跳跃性的方式进行传输,在一定程度上保护了参数信息的完整性,省去了中间繁琐的参数调整过程,极大地简化了学习目标和难度系数[31 ] .然而,可能正是这种残差学习方式,使得该模型没有学习到硅藻的关键形态学特征,间接导致其不能对涂片中的硅藻进行准确识别. ...
Fully convolutional neural network for detection and counting of diatoms on coatings after short-term field exposure
1
2020
... 在法医学自动化硅藻检验中,主要用到以CNN为核心的图片分类算法和目标检测算法.本研究结果发现,InceptionV3的各项评估指标(如查准率、召回率、F 1分数等)几乎均达到了90%或以上,如进行更大样本量的训练(如DiatomNet V1.0中的模型)其效能将会得到进一步提升.就本研究结果而言,InceptionV3模型的效能(F 1分数0.91,查准率92.58%,召回率89.89%,概率阈值0.5)均高于已发表的目标检测算法,如KRAUSE等[32 ] 的自建模型(F 1分数0.82,查准率77%,召回率87%)、RUIZ-SANTAQUITERIA等[33 ] 的Mask R-CNN(F 1分数0.85,查准率85%,召回率86%)、BUENO等[34 ] 的YOLO(F 1分数0.78,查准率75%,召回率84%).此外,本研究所采用的数字化WSI扫描技术能够在数分钟内获取整张涂片的数据,结合InceptionV3的高性能分类能力,使得DiatomNet V1.0无论在时间消耗、检测准确率、人工确认负担等方面均具有突出的优势,具有一定的实际应用前景.然而,不可否认的是,该系统主要基于传统光镜识别,放大倍数仅400倍,难以对硅藻种属进行准确判断.尽管ZHANG等[24 ] 的动物实验证实基于InceptionV3模型的硅藻种属分类方法能够大致推断出位于上海市黄浦江、苏州河水域的溺水地点,但为了获取更为精准的预测结果,仍需要基因测序、扫描电子显微镜等技术手段对硅藻种属进行更为细致的分类. ...
Semantic versus instance segmentation in microscopic algae detection
1
2020
... 在法医学自动化硅藻检验中,主要用到以CNN为核心的图片分类算法和目标检测算法.本研究结果发现,InceptionV3的各项评估指标(如查准率、召回率、F 1分数等)几乎均达到了90%或以上,如进行更大样本量的训练(如DiatomNet V1.0中的模型)其效能将会得到进一步提升.就本研究结果而言,InceptionV3模型的效能(F 1分数0.91,查准率92.58%,召回率89.89%,概率阈值0.5)均高于已发表的目标检测算法,如KRAUSE等[32 ] 的自建模型(F 1分数0.82,查准率77%,召回率87%)、RUIZ-SANTAQUITERIA等[33 ] 的Mask R-CNN(F 1分数0.85,查准率85%,召回率86%)、BUENO等[34 ] 的YOLO(F 1分数0.78,查准率75%,召回率84%).此外,本研究所采用的数字化WSI扫描技术能够在数分钟内获取整张涂片的数据,结合InceptionV3的高性能分类能力,使得DiatomNet V1.0无论在时间消耗、检测准确率、人工确认负担等方面均具有突出的优势,具有一定的实际应用前景.然而,不可否认的是,该系统主要基于传统光镜识别,放大倍数仅400倍,难以对硅藻种属进行准确判断.尽管ZHANG等[24 ] 的动物实验证实基于InceptionV3模型的硅藻种属分类方法能够大致推断出位于上海市黄浦江、苏州河水域的溺水地点,但为了获取更为精准的预测结果,仍需要基因测序、扫描电子显微镜等技术手段对硅藻种属进行更为细致的分类. ...
Lights and pitfalls of convolutional neural networks for diatom identification
1
2018
... 在法医学自动化硅藻检验中,主要用到以CNN为核心的图片分类算法和目标检测算法.本研究结果发现,InceptionV3的各项评估指标(如查准率、召回率、F 1分数等)几乎均达到了90%或以上,如进行更大样本量的训练(如DiatomNet V1.0中的模型)其效能将会得到进一步提升.就本研究结果而言,InceptionV3模型的效能(F 1分数0.91,查准率92.58%,召回率89.89%,概率阈值0.5)均高于已发表的目标检测算法,如KRAUSE等[32 ] 的自建模型(F 1分数0.82,查准率77%,召回率87%)、RUIZ-SANTAQUITERIA等[33 ] 的Mask R-CNN(F 1分数0.85,查准率85%,召回率86%)、BUENO等[34 ] 的YOLO(F 1分数0.78,查准率75%,召回率84%).此外,本研究所采用的数字化WSI扫描技术能够在数分钟内获取整张涂片的数据,结合InceptionV3的高性能分类能力,使得DiatomNet V1.0无论在时间消耗、检测准确率、人工确认负担等方面均具有突出的优势,具有一定的实际应用前景.然而,不可否认的是,该系统主要基于传统光镜识别,放大倍数仅400倍,难以对硅藻种属进行准确判断.尽管ZHANG等[24 ] 的动物实验证实基于InceptionV3模型的硅藻种属分类方法能够大致推断出位于上海市黄浦江、苏州河水域的溺水地点,但为了获取更为精准的预测结果,仍需要基因测序、扫描电子显微镜等技术手段对硅藻种属进行更为细致的分类. ...