近年来,随着法医遗传学的不断发展,亲缘关系鉴定的范围已由亲子鉴定扩展到更远、更复杂的关系,如全同胞、半同胞、叔侄、祖孙等复杂亲缘关系。复杂亲缘关系是指由于亲代个体的缺失,需要同胞、隔代或旁系亲属参与的亲缘关系鉴定。亲缘关系鉴定的应用范围极为广泛,特别是在司法鉴定领域,大多情况下不可能将生物样本的遗传资料与参考样本直接进行比较确定其身份时,如失踪人口及大规模灾难受害者的身份鉴定,往往需要通过与一个或多个可能的亲属进行亲缘关系鉴定间接获取其身份。此外,在移民、财产继承、寻亲、办理户籍等过程中也经常需要对两个体进行某一确定的亲缘关系鉴定。
一般而言,解决这类问题的方法依赖于似然比(likelihood ratio,LR)计算[1],其理论依据是血缘一致性(identity-by-descent,IBD)概率[2]。LR法又称ITO法,依据两个体间的IBD概率来计算叔侄关系指数,是亲缘关系鉴定的经典算法[3-4]。基于毛细管电泳(capillary electrophoresis,CE)的商品化试剂盒通常包含15~30个STR,具有较强的鉴别效能,能够满足亲子鉴定的需要[5-7]。然而,由于单个CE商品化试剂盒所包含的STR数量有限,往往不能满足一级及以上亲缘关系鉴定的需求[8]。研究[9-11]表明,基于二代测序平台的ForenSeqTM DNA Signature Prep试剂盒可同时检测27个常染色体STR、94个SNP、7个X-STR和24个Y-STR,能够有效地进行亲子关系及全同胞关系鉴定,但在二级亲缘关系鉴定中的系统效能相对较弱。ZHANG等[3]联合ForenSeqTM DNA Signature Prep试剂盒和AGCU 21+1 STR荧光检测试剂盒(包括40个STR和91个SNP),当以2和-2(lgLR)作为叔侄关系认定或排除的判定阈值时,系统效能为0.998 9。曹玉杰[12]基于二代测序平台的42个STR复合分型系统对真实家系样本二级亲缘关系研究中,当以1和-1(lgLR)作为叔侄、祖孙关系认定或排除的判定阈值时,系统效能为0.913 0。此外,陈慧等[13]基于CE商品化试剂盒,评估了不同阈值下在检测15~65个STR时9种常见的祖孙关系鉴定模型的系统效能,提示引入参考样本可有效地提高祖孙关系鉴定的系统效能,并提出全同胞是祖孙关系鉴定中优先选择的参考样本。这些研究表明,通过增加遗传标记数量或者引入参考样本可以有效提高复杂亲缘关系鉴定的系统效能。
在复杂亲缘关系中,叔侄关系是其中一种特殊的二级亲缘关系。广义的叔侄关系包括叔(姑)侄和舅(姨)甥。目前,叔侄关系鉴定的需求在司法鉴定领域越来越常见,但是尚无对应的技术标准及规范[10]。本研究为探究增加检测STR和引入参考样本对叔侄关系鉴定的影响,挑选了SiFaSTRTM 23plex身份鉴定系统、Goldeneye® DNA身份鉴定系统22NC和AGCU 21+1 STR荧光检测试剂盒这3种CE商品化试剂盒(包含55个非重复STR),基于华东汉族等位基因频率,针对引入不同的亲属构建5种常见的叔侄关系鉴定模型,分别模拟10 000对叔侄和10 000对对应的无关个体在检测19、39、55个STR时的LR,计算每种模型的灵敏度、特异性、系统效能及误判率等参数。此外,使用在华东地区汉族人群中采集的真实样本对模拟结果进行验证,进一步评估包含不同数量STR及引入不同已知亲属对系统效能的影响,为叔侄关系鉴定的法医学应用提供理论参考。
1 对象与方法
1.1 模拟实验设计
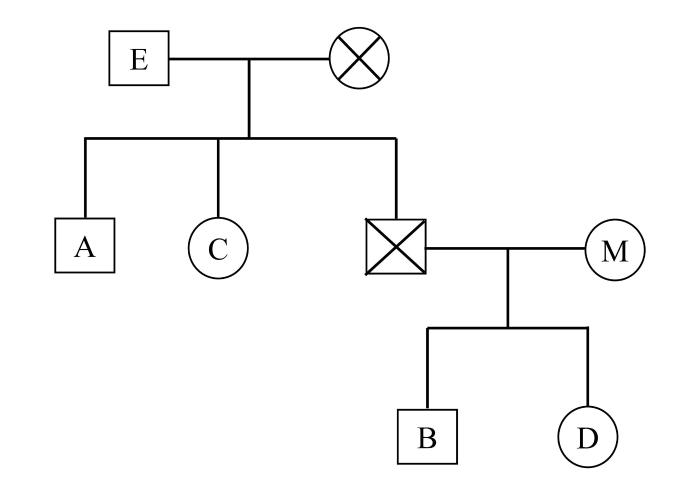
在一对存疑的叔(A)侄(B)关系鉴定中,除了叔侄(AB)两个体外,还可能存在其他明确的亲属,如姑(C)、全同胞(D)、祖辈(E)、生母(M)。个体间的亲缘关系如图1所示(忽视性别),针对引入不同的参考样本构建5种常见的叔侄关系鉴定模型:(1)叔侄两个体(AB);(2)引入A的全同胞C[(ABC)A];(3)引入A的父亲E[(ABE)A];(4)引入B的全同胞D[(ABD)B];(5)引入B的母亲M[(ABM)B]。
图1
图1
叔侄关系鉴定家系谱图
A:叔;B:侄;C:A的全同胞;D:B的全同胞;E:A的父亲;M:B的母亲。
Fig. 1
Pedigree chart of uncle-nephew relationship identification
1.2 模拟样本
假设STR之间相互独立,依据华东汉族等位基因频率[14-16]使用Familias v3.2软件(挪威Petter Mostad公司)[17-18]随机模拟上述5种常见的叔侄关系鉴定模型各10 000对和10 000对对应的无关个体在检测19、39、55个STR时的LR值。其中19个STR是SiFaSTRTM 23plex身份鉴定系统(司法鉴定科学研究院)包含的19个必检STR,39个STR是SiFaSTRTM 23plex身份鉴定系统和Goldeneye® DNA身份鉴定系统22NC[基点认知技术(北京)有限公司]去除重复后所包含的STR,55个STR是SiFaSTRTM 23plex身份鉴定系统、Goldeneye® DNA身份鉴定系统22NC和AGCU 21+1 STR荧光检测试剂盒(无锡中德美联生物技术有限公司)去除重复后所包含的STR。由于叔侄关系涉及的减数分裂次数较多,故突变模型设置为简单突变模型,其中男性突变率为0.002 0,女性突变率为0.000 5[13]。
1.3 验证
本研究主要集中分析叔侄关系与无关个体的甄别。为了评估模拟结果的实用价值,使用真实样本对其进行验证。在知情同意的情况下,采集来自浙江汉族9个家系85名志愿者的外周血,样本的采集与使用均已获得司法鉴定科学研究院伦理委员会的批准。使用QIAamp DNA Blood Mini试剂盒(德国Qiagen公司)提取DNA,基于CE平台使用上述3种试剂盒(SiFaSTRTM 23plex身份鉴定系统、Goldeneye® DNA身份鉴定系统22NC和AGCU 21+1 STR荧光检测试剂盒)对85个个体进行55个STR分型。基于之前研究中华东汉族群体等位基因频率[16],依据前期样本采集所收集的家系资料信息,参考《亲权鉴定技术规范》(GB/T 37223—2018)进行亲子鉴定,当累积亲权指数(parentage index,PI)≥10 000时,确认亲子关系。在浙江汉族9个家系85个个体中鉴定出71对亲子关系。依据亲子关系绘制家系图,确定叔侄关系对。共获得34对AB和175对无关个体,19对ABC与66对对应的无关个体,7对ABD与87对对应的无关个体,49对ABE与105对对应的无关个体,26对ABM与173对对应的无关个体。使用Familias v3.2软件计算各关系对在检测19、39、55个STR时的lgLR值,基于4种lgLR阈值(4与-4、3与-3、2与-2、1与-1)分别计算真实家系样本中检测19、39、55个STR时的相关参数,并与模拟结果进行比较。使用SPSS 22.0软件(美国IBM公司)对模拟结果与真实样本的计算结果进行配对t检验,检验水准α=0.05。
1.4 统计分析
2 结果
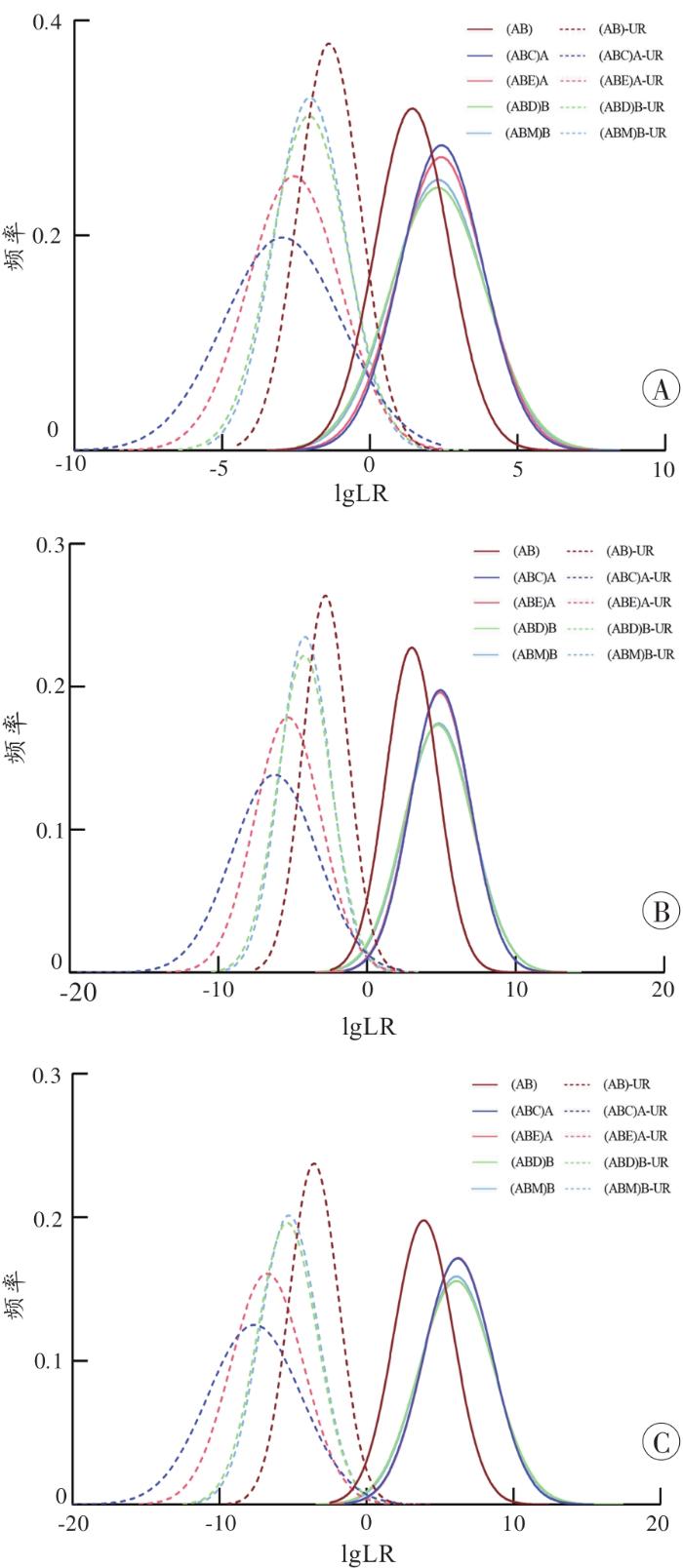
2.1 lgLR在模拟数据中的分布
图2
图2
5种叔侄关系鉴定模型在模拟数据上的lgLR分布图
A:19个STR;B:39个STR;C:55个STR。UR表示无关个体。
Fig. 2
Distribution of lgLR values of five models of uncle-nephew relationship identification in simulated data
表1 5种叔侄关系鉴定模型在19、39、55个STR检测体系的模拟数据和真实样本的lgLR均值
Tab. 1
| 来源 | (AB) | (AB)-UR | (ABC)A | (ABC)A-UR | (ABE)A | (ABE)A-UR | (ABD)B | (ABD)B-UR | (ABM)B | (ABM)B-UR |
|---|---|---|---|---|---|---|---|---|---|---|
| 模拟数据 | ||||||||||
| 55 STR | 3.970 3 | -3.539 8 | 6.240 5 | -7.856 0 | 6.261 6 | -6.756 5 | 6.229 0 | -5.321 1 | 6.182 7 | -5.196 9 |
| 39 STR | 3.091 3 | -2.755 0 | 4.951 5 | -6.433 6 | 4.951 4 | -5.312 8 | 4.887 7 | -4.186 5 | 4.895 6 | -4.084 6 |
| 19 STR | 1.516 3 | -1.339 3 | 2.449 8 | -3.184 9 | 2.439 1 | -2.565 4 | 2.410 0 | -2.012 2 | 2.380 6 | -1.960 0 |
| 真实样本 | ||||||||||
| 55 STR | 3.012 0 | -3.787 2 | 3.970 8 | -9.090 1 | 5.852 6 | -7.073 7 | 5.048 8 | -6.119 9 | 4.648 8 | -5.700 8 |
| 39 STR | 2.555 0 | -2.985 5 | 3.883 6 | -7.255 8 | 4.652 1 | -5.492 6 | 3.998 9 | -4.717 6 | 4.067 1 | -4.418 3 |
| 19 STR | 0.994 9 | -1.467 6 | 1.620 5 | -3.915 1 | 1.977 8 | -2.723 1 | 1.596 2 | -2.447 2 | 1.759 8 | -2.178 1 |
为了进一步明确增加检测STR数量和引入参考个体对叔侄关系鉴定模型的系统效能的改善,本研究计算了不同模型在4种lgLR阈值(4与-4、3与-3、2与-2、1与-1)下叔侄关系判定的灵敏度、特异性、系统效能及误判率等参数(表2)。当引入不同的参考个体时,(AB)、(ABC)A、(ABE)A、(ABD)B、(ABM)B模型在4种lgLR阈值(4与-4、3与-3、2与-2、1与-1)下检测19、39、55个STR时的灵敏度、特异性、系统效能及误判率见附表1。
随着检测STR数量的增加,灵敏度、特异性及系统效能随之增大,误判率则随之降低。如附表1所示,在同一检测体系下,5种模型的系统效能从小到大依次为:(AB)<(ABD)B≈(ABM)B<(ABC)A≈(ABE)A。可以看出,相对于引入其他亲属(D、M),引入叔A的父亲(母亲)E或全同胞C的系统效能更大。
2.2 真实样本的验证
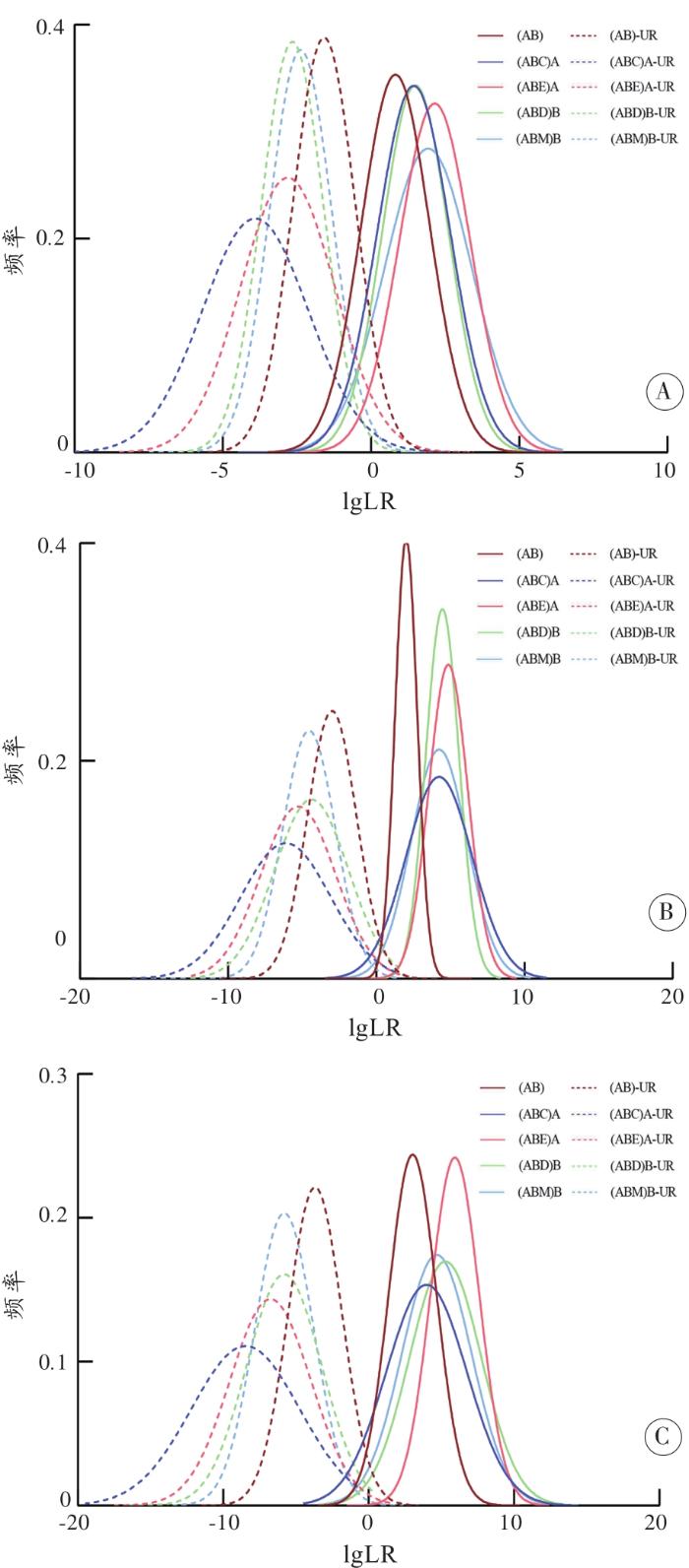
计算真实样本的lgLR值与模拟数据进行比较。当引入不同的参考个体时,真实家系样本中(AB)、(ABC)A、(ABE)A、(ABD)B、(ABM)B鉴定模型在检测55个STR时叔侄关系对的lgLR均值分别为3.012 0、3.970 8、5.852 6、5.048 8、4.648 8,对应的无关个体分别为-3.787 2、-9.090 1、-7.073 7、-6.119 9、 -5.700 8,叔侄关系对与无关个体对的重叠区域分别为4.78%、0、0、0、0(附表2)。经独立样本t检验,除(ABC)A模型外,其余模型真实样本在检测19、39、55个STR的lgLR值分布与对应的模拟数据之间差异无统计学意义(P>0.05),说明本研究使用的模拟数据是可靠的。
与模拟数据中的结果相一致,叔侄关系对的lgLR值均与对相应的无关个体对的lgLR值之间有差异,且随着检测STR数量的增加叔侄关系对的lgLR值增大,对应无关个体对的lgLR值减小;当检测相同数量的STR时,增加检测已知亲属的lgLR值均比两个体叔侄关系鉴定的lgLR值大(图3)。对于叔侄关系鉴定,当引入不同的参考个体时,5种模型(AB)、(ABC)A、(ABE)A、(ABD)B、(ABM)B在4种lgLR阈值(4与-4、3与-3、2与-2、1与-1)下检测19、39、55个STR时的灵敏度、特异性、系统效能及误判率见附表2。随着检测STR数量的增加,灵敏度、特异性及系统效能随之增大,误判率则随之降低。在同一检测体系下,真实样本中5种模型的系统效能与对应的模拟结果基本一致(配对样本t检验,P>0.05)。
图3
图3
5种叔侄关系鉴定模型在真实样本上的lgLR分布图
A:19个STR;B:39个STR;C:55个STR。UR表示无关个体。
Fig. 3
Distribution of lgLR values of five models of uncle-nephew relationship identification in true samples
3 讨论
目前,复杂亲缘关系鉴定的需求在法医鉴定中越来越常见,然而由于缺乏相应的技术标准(如叔侄关系鉴定规范),对于如何准确地推断个体间的复杂亲缘关系仍然是司法鉴定领域面临的重大挑战[3,9-11]。通常情况下,一方面利用已知亲属的常染色体STR分型,通过家系重建推断被鉴定人之间的亲缘关系[19];另一方面,也有研究[20-21]通过同时检测常染色体STR、性染色体STR及线粒体DNA更有效地确定亲缘关系。然而,在绝大多数情况下,很难采集到足够多的重要家庭成员用于家系重建,且检测性染色体STR或线粒体DNA往往不能提供有效的鉴定信息,因此,通过增加检测STR数和(或)引入已知亲属进行复杂亲缘关系鉴定是一种更加高效的鉴定方法。
前期研究[2,4,8]证实,增加遗传标记数量和引入参考样本均可以提高复杂亲缘关系鉴定的系统效能。本研究为了系统探讨增加检测STR数量和(或)引入已知亲属个体对叔侄关系鉴定的影响,通过模拟5种常见的叔侄关系鉴定模型,即叔侄两个体、2种引入叔(A)的亲属鉴定模型[(ABC)A、(ABE)A]和2种引入侄(B)的亲属鉴定模型[(ABD)B、(ABM)B],对19、39、55个STR检测系统的系统效能进行评估,并使用真实样本验证模拟结果的实用价值。由图2~3可见,在叔侄关系鉴定中,随着检测STR数量的增加,叔侄关系对和无关个体对的lgLR差异也越大,系统效能随着检测STR数量的增加而增强,其中包含55个STR的检测系统,在叔侄关系鉴定中的表现明显优于其他检测系统。此外,综合考虑灵敏度、特异性、系统效能、误判率等参数,当使用55个STR,以2和-2(lgLR)作为叔侄关系判定的阈值较为合适,这一阈值与美国血库协会(American Association of Blood Banks,AABB)[22]设置的LR>100作为亲缘关系判定的阈值相一致。当使用2和-2(lgLR)作为叔侄关系判定的阈值时(附表2),5种常见的叔侄关系鉴定模型的理论系统效能分别为0.830 4、0.973 4、0.970 0、0.950 6、0.951 0,误判率分别为0.11%、0.02%、0.03%、0.01%、0.05%;同样的,在真实样本中的系统效能分别为0.832 5、0.941 2、0.977 5、0.986 3、0.974 9,误判率均为0(可能与样本量较少有关)。当增加检测已知亲属个体时,不仅叔侄关系鉴定的系统效能均有不同程度的提高,误判率也随之下降,说明整体鉴定系统效能增加。如检测55个STR且以2和-2(lgLR)作为叔侄关系判定的阈值时,仅检测叔侄两个体的系统效能为0.830 4,误判率为0.11%,无法判断率为16.86%;当增加检测B的母亲的基因型时,仅检测39个STR时的系统效能为0.894 5,误判率为0.035%,无法判断率为10.52%。以上结果表明,引入已知亲属有时比增加检测STR数量更有效。真实样本的结果进一步证实了这一结论。依据本研究结果,建议首先考虑引用A的全同胞C、父亲(母亲)E,其次为引用B的全同胞D或母亲M。综上,增加检测STR数量和引入参考个体是提高叔侄关系鉴定模型系统效能的有效方法。
对于其他类型的二级亲缘关系(如祖孙、半同胞),增加检测常染色体STR和引入参考样本均有可能增强亲缘关系鉴定的系统效能,但最佳的参考样本有可能不同。由于不同的二级亲缘关系具有相同的IBD概率,仅检测相互独立的常染色体STR不能将其区分,根据具体案例可通过检测性染色体遗传标记、线粒体DNA等进行辅助甄别。增加检测常染色体STR和引入参考样本对其他二级亲缘关系的影响及二级亲缘关系间的区分将在后续的研究中进一步探讨。
在本研究中,通过比较叔侄关系鉴定的常见模型,评估包含不同数量STR、增加检测不同已知亲属以及不同阈值对系统效能的影响,为叔侄关系鉴定的法医学应用提供理论参考。在叔侄关系鉴定中,主要基于常染色体STR进行研究,未考虑性染色体STR及线粒体DNA。当检测常染色体STR基因座无法给出倾向性意见时,可依据父系或母系遗传特点,增加检测X-STR、Y-STR及mtDNA作为辅助进一步分析判定,从而提高鉴定的准确率,降低误判风险。
参考文献
ISFG: Recommendations on biostatistics in paternity testing
[J].
Genetic relatedness analysis: Modern data and new challenges
[J].
Pairwise kinship testing with a combination of STR and SNP loci
[J].
Optimizing the information increase through the addition of relatives and genetic markers in identification and kinship cases
[J].
Determination of the sensitivity and specificity of sibship calculations using AmpF lSTR Profiler Plus
[J].
Developmental validation of the GlobalFiler® Express PCR Amplification Kit: A 6-dye multiplex assay for the direct amplification of reference samples
[J].
Front cover: Development and validation of a novel SiFaSTRTM 23-plex system
[J].
Evaluation of advanced multiplex short tandem repeat systems in pairwise kinship analysis
[J].
Improved pairwise kinship analysis using massively parallel sequencing
[J].
Utility of ForenSeq™ DNA Signature Prep Kit in the research of pairwise 2nd-degree kinship identification
[J].
Pairwise kinship analysis of 17 pedigrees using massively parallel sequencing
[J].
NGS-STR体系在二级亲缘关系鉴定中的法医学应用研究
[D].
Forensic application research of NGS-STR system in 2nd-degree kinship identification
[D].
祖孙关系鉴定中引入不同参考个体的系统效能分析
[J].
Efficiency evaluation of grandparent-grandchild kinship analysis introducing different relative references
[J].
GoldeneyeTM DNA身份鉴定系统22NC试剂盒的法医遗传学调查
[J].
Forensic investigation of GoldeneyeTM DNA ID 22NC kit
[J].
21个非CODIS STR基因座的遗传多态性
[J].
Genetic polymorphisms of 21 non-CODIS STR loci
[J].
SiFaSTRTM 23plex DNA身份鉴定系统在华东汉族人群中的法医学应用
[J].
Forensic application of SiFaSTRTM 23plex DNA ID system in Han population of Eastern China
[J].
Familias 3 - Extensions and new functionality
[J].
多系统联合分析应用于复杂亲缘关系鉴定
[J].
Complex kinship analysis by multi-genotyping system and IBS score
[J].
Interest of X chromosome (Argus X-12 kit) in complex kinship analysis
[J].
Potentials and limits of pairwise kinship analysis using autosomal short tandem repeat loci
[J].
Annual report summary for testing in 2010
[R/OL]. (
The estimation of map distances from recombination values
[J].
The recombination landscape around forensic STRs: Accurate measurement of genetic distances between syntenic STR pairs using HapMap high density SNP data
[J].
Evaluation of the impact of genetic linkage in forensic identity and relationship testing for expanded DNA marker sets
[J].