肉及肉制品真实性鉴别技术的研究进展
2
2022
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
... DNA作为生物体的主要遗传物质,具有较好的结构稳定性及遗传信息的物种特异性,是用于种属鉴定的理想生物大分子[1].其中,线粒体DNA(mitochondrial DNA,mtDNA)因其自身的独特优势,在物种鉴定中具有较为可观的研究前景.mtDNA是细胞中独立于细胞核DNA之外的遗传物质,通常以多拷贝形式存在,相较于细胞核DNA更易于从组织细胞中分离而获取,可以满足法医微量检材的检测需求,在无法检测细胞核DNA的陈旧、降解、腐败甚至是烹煮过的检材中具有较大的应用潜力[8-10].更重要的是,mtDNA进化速率较快,约为细胞核DNA的5~10倍,既具有物种间的差异性,又具有物种内的遗传稳定性[11],是进行种属鉴定、研究近缘种属关系及进化的良好分子标记. ...
Research progress of meat and meat products authenticity identification technology
2
2022
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
... DNA作为生物体的主要遗传物质,具有较好的结构稳定性及遗传信息的物种特异性,是用于种属鉴定的理想生物大分子[1].其中,线粒体DNA(mitochondrial DNA,mtDNA)因其自身的独特优势,在物种鉴定中具有较为可观的研究前景.mtDNA是细胞中独立于细胞核DNA之外的遗传物质,通常以多拷贝形式存在,相较于细胞核DNA更易于从组织细胞中分离而获取,可以满足法医微量检材的检测需求,在无法检测细胞核DNA的陈旧、降解、腐败甚至是烹煮过的检材中具有较大的应用潜力[8-10].更重要的是,mtDNA进化速率较快,约为细胞核DNA的5~10倍,既具有物种间的差异性,又具有物种内的遗传稳定性[11],是进行种属鉴定、研究近缘种属关系及进化的良好分子标记. ...
Design a highly specific sequence for electrochemical evaluation of meat adulteration in cooked sausages
1
2020
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
Meat food fraud risk in Chinese markets 2012-2021
1
2023
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
189-z
1
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
基于DNA条形码技术常见肉类掺假鉴别技术的研究
1
2016
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
Techniques for identifying common meat adulterations based on DNA barcoding
1
2016
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
A rapid and reliable multiplex PCR assay for simultaneous detection of fourteen animal species in two tubes
2
2019
... 肉类食品掺假已成为一个全球性问题,尽管各国食品安全部门对肉及肉制品生产者和经营者具有明确的法律法规约束[1-2],但由于不同肉类价格差异带来的经济效益诱惑,不法分子仍会用猪肉、鸡肉、鸭肉等廉价肉类来替代牛肉、羊肉这些高价肉类进行贩卖和销售[3-4],加之我国肉类交易市场分布零散、监管盲点较多,使得肉类掺假问题层出不穷.肉类掺假不仅破坏市场秩序,导致经济损失,还可能影响消费者的健康,甚至引发宗教冲突[5].同时我国是肉类生产和消费大国,肉类食品的真实性和可追溯性是最重要的社会问题之一,关乎经济、公共安全、宗教信仰、生态安全、食品安全等.因此,对肉及肉制品进行种属鉴定是法庭科学工作者的重要任务,也是法医物证领域亟待解决的难题之一. ...
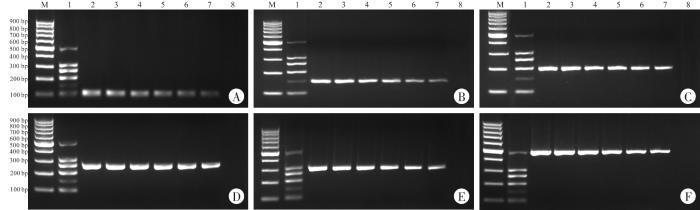
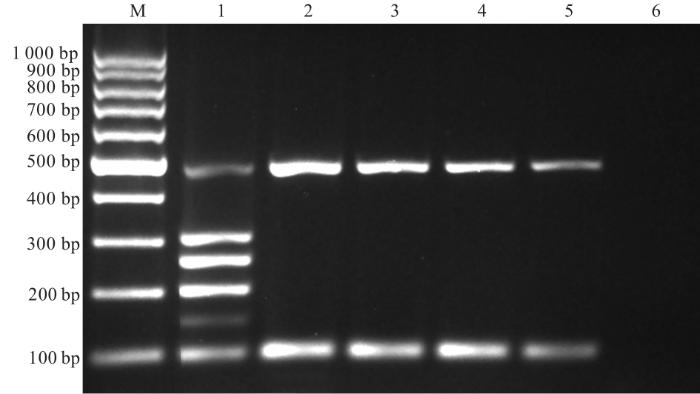
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...
DNA分析与种属鉴定
1
2003
... 在肉类掺假的检测和鉴定中,传统方法主要包括形态学检测法、组织学检测法、血清学检测法[6]等.然而,上述方法对样本质量要求较高,需要样本、组织保存完整.此外,上述鉴定方法特异性不足,尤其是血清学检测法,因受蛋白质稳定性的制约,难以应用于被高温破坏和降解检材的鉴定,同时组织学和形态学检测方法还对检验人员的经验要求较高,鉴定意见的主观性强,故存在较大的局限性[7].因此,为了准确、高效地检测肉及肉制品的种属来源,保护消费者免受不法行为的侵害,亟须建立一种简单、快速、准确的肉类种属鉴定方法. ...
DNA-based technique and species identification
1
2003
... 在肉类掺假的检测和鉴定中,传统方法主要包括形态学检测法、组织学检测法、血清学检测法[6]等.然而,上述方法对样本质量要求较高,需要样本、组织保存完整.此外,上述鉴定方法特异性不足,尤其是血清学检测法,因受蛋白质稳定性的制约,难以应用于被高温破坏和降解检材的鉴定,同时组织学和形态学检测方法还对检验人员的经验要求较高,鉴定意见的主观性强,故存在较大的局限性[7].因此,为了准确、高效地检测肉及肉制品的种属来源,保护消费者免受不法行为的侵害,亟须建立一种简单、快速、准确的肉类种属鉴定方法. ...
DNA分析在法医学种属鉴定中的应用研究
1
2006
... 在肉类掺假的检测和鉴定中,传统方法主要包括形态学检测法、组织学检测法、血清学检测法[6]等.然而,上述方法对样本质量要求较高,需要样本、组织保存完整.此外,上述鉴定方法特异性不足,尤其是血清学检测法,因受蛋白质稳定性的制约,难以应用于被高温破坏和降解检材的鉴定,同时组织学和形态学检测方法还对检验人员的经验要求较高,鉴定意见的主观性强,故存在较大的局限性[7].因此,为了准确、高效地检测肉及肉制品的种属来源,保护消费者免受不法行为的侵害,亟须建立一种简单、快速、准确的肉类种属鉴定方法. ...
A study of DNA analysis in forensic species identification
1
2006
... 在肉类掺假的检测和鉴定中,传统方法主要包括形态学检测法、组织学检测法、血清学检测法[6]等.然而,上述方法对样本质量要求较高,需要样本、组织保存完整.此外,上述鉴定方法特异性不足,尤其是血清学检测法,因受蛋白质稳定性的制约,难以应用于被高温破坏和降解检材的鉴定,同时组织学和形态学检测方法还对检验人员的经验要求较高,鉴定意见的主观性强,故存在较大的局限性[7].因此,为了准确、高效地检测肉及肉制品的种属来源,保护消费者免受不法行为的侵害,亟须建立一种简单、快速、准确的肉类种属鉴定方法. ...
Multiplex amplification of mitochondrial DNA for human and species identification in forensic evaluation
1
1999
... DNA作为生物体的主要遗传物质,具有较好的结构稳定性及遗传信息的物种特异性,是用于种属鉴定的理想生物大分子[1].其中,线粒体DNA(mitochondrial DNA,mtDNA)因其自身的独特优势,在物种鉴定中具有较为可观的研究前景.mtDNA是细胞中独立于细胞核DNA之外的遗传物质,通常以多拷贝形式存在,相较于细胞核DNA更易于从组织细胞中分离而获取,可以满足法医微量检材的检测需求,在无法检测细胞核DNA的陈旧、降解、腐败甚至是烹煮过的检材中具有较大的应用潜力[8-10].更重要的是,mtDNA进化速率较快,约为细胞核DNA的5~10倍,既具有物种间的差异性,又具有物种内的遗传稳定性[11],是进行种属鉴定、研究近缘种属关系及进化的良好分子标记. ...
Sequence analysis of mitochondrial 16S rRNA gene to identify meat species
0
2013
Evaluation of the suitability of mitochondrial DNA for species identification of microtraces and forensic traces
1
2017
... DNA作为生物体的主要遗传物质,具有较好的结构稳定性及遗传信息的物种特异性,是用于种属鉴定的理想生物大分子[1].其中,线粒体DNA(mitochondrial DNA,mtDNA)因其自身的独特优势,在物种鉴定中具有较为可观的研究前景.mtDNA是细胞中独立于细胞核DNA之外的遗传物质,通常以多拷贝形式存在,相较于细胞核DNA更易于从组织细胞中分离而获取,可以满足法医微量检材的检测需求,在无法检测细胞核DNA的陈旧、降解、腐败甚至是烹煮过的检材中具有较大的应用潜力[8-10].更重要的是,mtDNA进化速率较快,约为细胞核DNA的5~10倍,既具有物种间的差异性,又具有物种内的遗传稳定性[11],是进行种属鉴定、研究近缘种属关系及进化的良好分子标记. ...
Evolution of animal mitochondrial DNA: Relevance for population biology and systematics
1
1987
... DNA作为生物体的主要遗传物质,具有较好的结构稳定性及遗传信息的物种特异性,是用于种属鉴定的理想生物大分子[1].其中,线粒体DNA(mitochondrial DNA,mtDNA)因其自身的独特优势,在物种鉴定中具有较为可观的研究前景.mtDNA是细胞中独立于细胞核DNA之外的遗传物质,通常以多拷贝形式存在,相较于细胞核DNA更易于从组织细胞中分离而获取,可以满足法医微量检材的检测需求,在无法检测细胞核DNA的陈旧、降解、腐败甚至是烹煮过的检材中具有较大的应用潜力[8-10].更重要的是,mtDNA进化速率较快,约为细胞核DNA的5~10倍,既具有物种间的差异性,又具有物种内的遗传稳定性[11],是进行种属鉴定、研究近缘种属关系及进化的良好分子标记. ...
Development and validation of a novel five-dye short tandem repeat panel for forensic identification of 11 species
1
2020
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Species identification of fox-, mink-, dog-, and rabbit-derived ingredients by multiplex PCR and real-time PCR assay
1
2018
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Meat species identification by two direct-triplex real-time PCR assays using low resolution melting
1
2017
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Multiplex PCR and 12S rRNA gene sequencing for detection of meat adulteration: A case study in the Egyptian markets
2
2021
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...
Droplet digital polymerase chain reaction (ddPCR) assays integrated with an internal control for quantification of bovine, porcine, chicken and turkey species in food and feed
1
2017
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
PCR-RFLP authentication of meats from red Deer (Cervus elaphus), fallow Deer (Dama dama), Roe Deer (Capreolus capreolus), cattle (Bos taurus), sheep (Ovis aries), and goat (Capra hircus)
1
2006
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
PCR-RFLP for authentication of different species of processed snappers using mitochondrial D-loop region by single enzyme
1
2018
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Development of a simultaneous identification method for 13 animal species using two multiplex real-time PCR assays and melting curve analysis
2
2018
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...
A sensitive multiplex PCR protocol for simultaneous detection of chicken, duck, and pork in beef samples
1
2019
... 肉类掺假一直以来都是食品安全领域备受关注的重要问题,掺杂其他肉类或添加非肉类成分不仅欺骗消费者,还严重影响人体健康[20-21].因此,准确鉴定肉类成分在食品安全领域具有重要意义. ...
A multiplex PCR method mediated by universal primers for the identification of eight meat ingredients in food products
2
2019
... 肉类掺假一直以来都是食品安全领域备受关注的重要问题,掺杂其他肉类或添加非肉类成分不仅欺骗消费者,还严重影响人体健康[20-21].因此,准确鉴定肉类成分在食品安全领域具有重要意义. ...
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...
A multiplex assay to identify 18 European mammal species from mixtures using the mitochondrial cytochrome b gene
0
2008
肉制品中狗、狐、貂源性成分DNA检测试剂盒的制备
0
2021
Development of DNA detection kit for dog, fox and mink-derived components in adulterated meat products
0
2021
应用16S rRNA基因测序鉴定肉类掺假1例
1
2022
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Identification of a case of meat adulteration by 16S rRNA gene sequencing
1
2022
... 近年来,随着分子生物学技术的快速发展,基于PCR的DNA检测技术被广泛应用于肉及肉制品的种属鉴定,并被证明是一种潜在、高效和灵敏的检测技术[12].目前,应用于肉及肉制品种属鉴定的PCR技术主要包括常规PCR、实时荧光定量PCR[13-14]、多重PCR[15]、数字PCR[16]、限制性片段长度多态性(restriction fragment length polymorphism,RFLP)[17-18]等.与传统的肉类掺假鉴定技术相比,这些基于PCR的DNA检测技术由于准确性高、灵敏度高、特异性强、可追溯性强等优势,已成为肉类掺假鉴定的主要技术手段[19-24].其中,多重PCR是一种简单、高效、低成本的技术,通过使用多对特异性引物同时扩增目标DNA序列,可以在同一反应体系中鉴别多个物种,采用简单的琼脂糖凝胶电泳即可进行分析,既节省时间及成本,又可提供更准确的鉴定意见. ...
Sequence analysis of mitochondrial 12S rRNA gene can identify meat species
1
2004
... 使用12S rRNA基因的通用引物[25],对采集的样本DNA进行扩增并对扩增产物进行双向Sanger测序,将测序序列校对、拼接后于美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)基于局部比对算法的搜索工具(Basic Local Alignment Search Tool,BLAST)进行比对,以验证所采集样本的准确性.测序由生工生物工程(上海)股份有限公司完成. ...
MEGA11:Molecular evolutionary genetics analysis version 11
1
2021
... 从GenBank数据库(www.ncbi.nlm.nih.gov)中下载常见食用肉类物种牛(NC_006853.1)、羊(NC_001941.1、NC_005044.2)、鸡(NC_053523.1)、猪(NC_000845.1)、鹅(NC_011196.1)、鸭(NC_009684.1)的线粒体全基因组序列.使用MEGA 11软件[26]筛选具有种内保守性、种间特异性的DNA序列作为物种特异性DNA序列.物种特异性DNA序列纳入标准:(1)该序列在不同物种之间具有足够的碱基差异,特别是近缘物种之间;(2)该序列在分布于不同地理区域的同一物种内没有碱基长度差异;(3)数据库中关于该序列的资料全面,能够查找到该序列不同物种的信息;(4)该序列长度大于200 bp,便于引物的设计.基于物理参数(如解链温度、二级结构、引物错配性等),采用Primer Premier 5.0软件(加拿大Premier公司)设计6对物种特异性引物,并使用NCBI的BLAST评估所设计引物的特异性.利用AutoDimer 1.0软件[27]模拟多重PCR反应,以确保在多重PCR体系中不会发生引物间交叉反应.引物序列及扩增产物长度见表1,引物均由生工生物工程(上海)股份有限公司合成. ...
AutoDimer: A screening tool for primer-dimer and hairpin structures
1
2004
... 从GenBank数据库(www.ncbi.nlm.nih.gov)中下载常见食用肉类物种牛(NC_006853.1)、羊(NC_001941.1、NC_005044.2)、鸡(NC_053523.1)、猪(NC_000845.1)、鹅(NC_011196.1)、鸭(NC_009684.1)的线粒体全基因组序列.使用MEGA 11软件[26]筛选具有种内保守性、种间特异性的DNA序列作为物种特异性DNA序列.物种特异性DNA序列纳入标准:(1)该序列在不同物种之间具有足够的碱基差异,特别是近缘物种之间;(2)该序列在分布于不同地理区域的同一物种内没有碱基长度差异;(3)数据库中关于该序列的资料全面,能够查找到该序列不同物种的信息;(4)该序列长度大于200 bp,便于引物的设计.基于物理参数(如解链温度、二级结构、引物错配性等),采用Primer Premier 5.0软件(加拿大Premier公司)设计6对物种特异性引物,并使用NCBI的BLAST评估所设计引物的特异性.利用AutoDimer 1.0软件[27]模拟多重PCR反应,以确保在多重PCR体系中不会发生引物间交叉反应.引物序列及扩增产物长度见表1,引物均由生工生物工程(上海)股份有限公司合成. ...
Species authentication of common meat based on PCR analysis of the mitochondrial COⅠ gene
2
2015
... Information on specific primers for six common edible meat species
Tab. 1| 物种 | 目标基因 | 引物序列(5´→3´) | 扩增长度/bp | 引物浓度/(μmol·L-1) |
|---|
| 牛 | 16S rRNA | 正向:ACACAGGAGTGCATCTAAGG | 104 | 0.40 |
| 反向:AATACTGGGAATGCTGGAGG |
| 羊 | COX-1[28] | 正向:CACGACGATACTCTGATTAC | 157 | 0.40 |
| 反向:GTGGTTAGGTCTACAGTTAG |
| 鸡 | Cytb | 正向:ACACTTGCCGGAACGTACAA | 205 | 0.16 |
| 反向:CCCCATGGGAGAACATAGCC |
| 猪 | COX-1[28] | 正向:CGGGTACACACTCAACCAAG | 268 | 0.16 |
| 反向:TGTGCTTGTCAGTTCTACTGC |
| 鹅 | NADH2 | 正向:TGCTACTCTCGACCCTCAT | 313 | 0.32 |
| 反向:GTTGTGATCATGGATAGGTTAAGG |
| 鸭 | 16S rRNA | 正向:CTAGCTCAGCCGCTTAAACA | 495 | 0.56 |
| 反向:TCCACTAGTCGAGGTTGTGT |
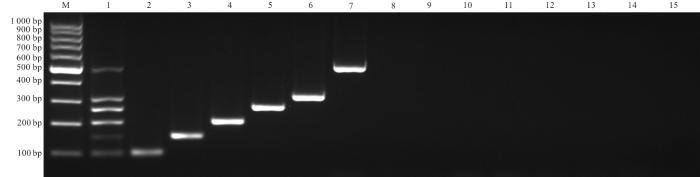
1.4 单一和多重PCR检测基于新设计的物种特异性引物对每个目标物种的样本DNA进行单一PCR扩增.PCR体系总体积为20 μL,10 μL 2×PCR预混液,8 μL无核酶水,1 μL正、反向引物混合物,1 μL DNA(1 ng/μL).初始扩增条件:94 ℃ 5 min;95 ℃ 30 s,57 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min.PCR扩增产物经2%琼脂糖凝胶电泳检测,以100 bp DNA Ladder(苏州近岸蛋白公司)为分子量标准,使用E-GelTM Power Snap核酸电泳系统(美国Thermo Fisher Scientific公司)分析扩增产物. ...
... [
28]
正向:CGGGTACACACTCAACCAAG | 268 | 0.16 | | 反向:TGTGCTTGTCAGTTCTACTGC |
| 鹅 | NADH2 | 正向:TGCTACTCTCGACCCTCAT | 313 | 0.32 |
| 反向:GTTGTGATCATGGATAGGTTAAGG |
| 鸭 | 16S rRNA | 正向:CTAGCTCAGCCGCTTAAACA | 495 | 0.56 |
| 反向:TCCACTAGTCGAGGTTGTGT |
1.4 单一和多重PCR检测基于新设计的物种特异性引物对每个目标物种的样本DNA进行单一PCR扩增.PCR体系总体积为20 μL,10 μL 2×PCR预混液,8 μL无核酶水,1 μL正、反向引物混合物,1 μL DNA(1 ng/μL).初始扩增条件:94 ℃ 5 min;95 ℃ 30 s,57 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min.PCR扩增产物经2%琼脂糖凝胶电泳检测,以100 bp DNA Ladder(苏州近岸蛋白公司)为分子量标准,使用E-GelTM Power Snap核酸电泳系统(美国Thermo Fisher Scientific公司)分析扩增产物. ...
一种利用线粒体DNA鉴别羊肉、猪肉、鸡肉、鸭肉的PCR检测方法
1
2013
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...
A PCR-mtDNA approach for the identification of lamb, pork, chicken and duck
1
2013
... 基于DNA的多重PCR检测技术是一种快速、准确、特异、灵敏且易于操作的肉类掺假鉴定方法,该方法通过在同一体系中同时扩增多对物种特异性引物,从而达到快速鉴别肉类食品中不同物种成分的目的.然而,多重PCR体系中引物数量的增加会导致更多的交叉反应[5,21],这些交叉反应是限制同一体系中可鉴定物种数量的主要原因.此外,多重PCR体系中,由于不同引物与模板DNA处于同一反应体系,导致引物与模板之间更容易发生非特异性扩增.因此,本研究基于线粒体全基因组序列严格按照特异性DNA序列筛选标准,选择具有种内保守性、种间特异性的物种特异性DNA序列用于引物设计,以提高多重PCR检测体系的特异性和可靠性.但需要注意的是,在某些加工肉制品中,DNA可能会发生降解导致PCR扩增失败.研究[15,29]表明,当肉加热到100 ℃时DNA片段长度减少至1 100 bp左右,加热到120 ℃时则减少至600 bp以下.因此,为提高PCR扩增成功率,本研究特异性引物所扩增的目标片段均在500 bp以下,并且成功扩增出6种目标肉类的特异性条带,在1 000 bp内未产生非特异性扩增片段.同时,将所构建的多重PCR检测体系对驴、狗、兔、马、鼠、猫、人7个常见物种的DNA样本进行扩增,结果未出现非特异性条带,表明该体系具有良好的种属特异性.在多重PCR反应体系中,多重PCR反应的灵敏度通常比单个PCR反应低10~100倍[19],这种灵敏度的降低可能是由于多对引物之间的交叉反应和引物与模板DNA之间的非特异性结合所导致.尽管如此,本研究建立的多重PCR检测体系能够检测到的DNA量低至0.062 5 ng,这一灵敏度水平足以用于实际案件. ...